Une révolution dans l’imagerie des cellules végétales
En 2015, une nouvelle technique de microscopie, appelée microscopie d’expansion, a bouleversé l’imagerie biologique en dépassant les limites de résolution des systèmes classiques et en rendant accessible la microscopie de super-résolution à moindre coût. Cependant, son application aux tissus végétaux multicellulaires et rigides restait un défi méthodologique. Dans un article publié dans la revue Plant Cell, des scientifiques développent une méthode, appelée ROOT-ExM, qui permet, 10 ans après, de relever ce défi.
La microcopie d’expansion : une avancée majeure en microscopie photonique
La microscopie d’expansion (ExM), introduite en 2015 par l’équipe d’Ed Boyen a bouleversé l’imagerie biologique. Cette technique permet d’agrandir physiquement les échantillons, dépassant ainsi la limite de résolution des systèmes d’acquisition photoniques classiques et offrant une visualisation des événements cellulaires à l’échelle du nanomètre (soit un millionième de millimètre !). En plus d'offrir une résolution subcellulaire importante, l’Expansion est une solution économique offrant une alternative abordable aux méthodes de super-résolution nécessitant des instruments très coûteux. L’Expansion permet désormais d’accéder à la microscopie de super-résolution avec un équipement standard, rendant cette technologie plus accessible à la communauté scientifique.
ROOT-ExM : une méthode pensée pour les plantes
Bien que largement adoptée pour divers échantillons biologiques, son application aux tissus végétaux restait rare, faute d’une méthodologie adaptée, malgré une forte demande scientifique. Dans une étude publiée dans Plant Cell, les scientifiques ont développé ROOT-ExM, une méthode d’expansion spécifiquement conçue pour les tissus végétaux multicellulaires rigides (car entourés d'une paroi extracellulaire) et complexes. ROOT-ExM permet une expansion isotrope de l’échantillon par un facteur quatre. Cela signifie que l’expansion est uniforme dans toutes les directions. En pratique, cela garantit que les structures internes des échantillons conservent leur proportion d’origine après agrandissement.
La méthode ROOT-ExM commence tout d’abord avec une étape de marquage. Les scientifiques peuvent utiliser des anticorps spécifiques, des colorants capables de cibler certains compartiments cellulaires, ou encore préserver la fluorescence native des protéines. L’échantillon est ensuite ancré et inclus dans un gel qui, après digestion enzymatique, se dilate de façon isotrope au contact de l’eau. ROOT-ExM a été utilisé avec succès pour imager une variété d’organites et de compartiments subcellulaires, comme l’appareil de Golgi, le réticulum endoplasmique et le cytosquelette, révélant des détails inaccessibles par des méthodes classiques. Associée à des techniques de super résolution telles que la microscopie de déplétion par émission stimulée (STED), ROOT-ExM permet d’atteindre une résolution de 20 nm et d’imager les plasmodesmes, des canaux membranaires nanoscopiques enchâssés dans la paroi. Lorsqu’elle est combinée à la microscopie à feuillet de lumière (LLSM), ROOT-ExM permet une analyse 3D précise et isotropique des processus cellulaires à l'échelle nanométrique, comme la quantification des vésicules lors de la division cellulaire.
L’introduction de ROOT-ExM marque un tournant majeur en biologie végétale, en proposant une solution robuste et accessible pour l’imagerie de super-résolution. Cette avancée ouvre de nouvelles perspectives pour l'exploration de l’architecture subcellulaire des plantes. Cette avancée répond à un besoin crucial dans la communauté scientifique, qui réclamait depuis longtemps des techniques plus performantes pour l’étude des tissus végétaux à l’échelle nanométrique.
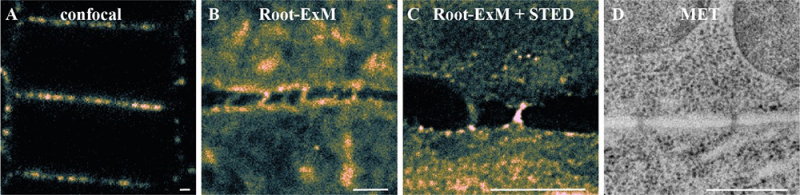
Figure : Les plasmodesmes sont des canaux de communication nanométriques (20 nm de diamètre pour 150 de long) qui traversent la paroi des cellules végétales et que l’on perçoit en microscopie conventionnelle comme un simple « point » (confocal, chaque point est un ou plusieurs plasmodesmes). Après expansion de l’échantillon avec le protocole Root-ExM les plasmodesmes peuvent être facilement visualisés en microscopie confocale (Root-ExM). Combinée au STED, Root-ExM permet d’identifier très clairement les plasmodesmes qui traversent la paroi des cellules végétales. Échelle : A, B et C 1 µm, D 0.5µm
En savoir plus : Magali S Grison, Guillaume Maucort, Amandine Dumazel, Dorian Champelovier, Yutaro Shimizu, Yohann Boutté, Mónica Fernández-Monreal, Emmanuelle M Bayer, Root expansion microscopy: A robust method for super resolution imaging in Arabidopsis, The Plant Cell, Volume 37, Issue 4, April 2025, koaf050, https://doi.org/10.1093/plcell/koaf050
Contact
Laboratoire
Laboratoire de biogenèse membranaire - LBM (CNRS/Université de Bordeaux)
71 avenue Edouard Bourlaux
33140 Villenave d'Ornon
