Réparation de l’ADN : comment retrouver la séquence identique dans l’immensité du génome
Les cassures de l’ADN sont des lésions toxiques pour la cellule. Afin de réparer ces cassures il est nécessaire, à partir de l’extrémité de ces cassures, de retrouver une séquence homologue dans l’immensité du génome. Dans un article publié dans Molecular Cell des scientifiques décrivent un modèle qui permet d’expliquer cette pêche à la séquence homologue.
La stabilité du matériel génétique est essentielle au bon fonctionnement de chacune de nos cellules. Néanmoins des accidents peuvent se produire, par exemple sous l’effet de radiations ou d’erreurs lors de la réplication de l’ADN. Pour réparer ces cassures il existe des mécanismes extrêmement conservés chez toutes les espèces qui ont à charge d’opérer une réparation fidèle. La recombinaison homologue est une voie de réparation à haute-fidélité des cassures qui utilise une molécule d’ADN intacte de séquence identique comme matrice. Cette séquence peut provenir de la chromatide sœur, du chromosome homologue plus distant, ou d’autres séquences répétées dans le génome. Cette copie homologue intacte doit donc être identifiée dans l’immensité du génome et de l’espace nucléaire. Comme est conduite cette recherche d’une aiguille homologue dans la botte de foin du génome ?
Pour répondre à cette question, des scientifiques du CNRS ont développé une technique de génomique à haut-débit appelée ssHi-C chez la levure Saccharomyces cerevisiae, un modèle pour l’étude des mécanismes fondamentaux d’entretien de l’ADN. Cette technique leur a permis de cartographier à l’échelle du génome les contacts faits par la cassure. Ces données ont permis de révéler que la recherche d’homologie procédait en deux grandes phases :
- une première phase au sein de laquelle la structuration du génome par les cohésines contraint la recherche localement, les cohésines étant des protéines qui assurent la cohésion des chromatides sœurs en piégeant l’ADN en leur sein.
- une seconde phase au cours de laquelle la recherche s’émancipe de ces contraintes et interroge des sites génomiques distants. Cette recherche à longue portée est permise par la production de longs ADN simple-brins de part et d’autre de la cassure, et de leur rigidification par la formation d’un filament de la recombinase Rad51. Cette protéine, qui catalyse les événements de recombinaison, agit dans ce cas telle une canne à pêche et projette sa ligne constituée de longs ADN simple-brins loin dans le noyau. Les deux extrémités de la cassure cherchent de manière coordonnée et si une séquence homologue est trouvée par une extrémité de la cassure, alors l’autre extrémité cherche de manière accrue dans son voisinage. De manière surprenante, certaines régions du génome engagent la cassure plus fréquemment que d’autres, tel le chromosome III. Le génome n’est donc pas homogènement interrogé lors de la recherche d’homologie, ce qui suggère l’existence de mécanismes de ciblage qui feront l’objet d’études ultérieures.
Ces découvertes conduisent à un modèle général de la recherche d’homologie basé sur la structure rigide du filament de Rad51 et ses partenaires. Elle ouvre de nouvelles directions de recherche, notamment pour comprendre les défauts associés à des mutations dans des gènes suppresseurs de tumeurs, tels que les paralogues de Rad51, dans la régulation de la structure du filament de Rad51 et la portée de la recherche d’homologie. Finalement, la technique du ssHi-C, universellement applicable, devrait permettre d’étudier la localisation spatiale de l’ADN au cours de la réplication ou de sa réparation dans de nombreux organismes et contextes cellulaires.
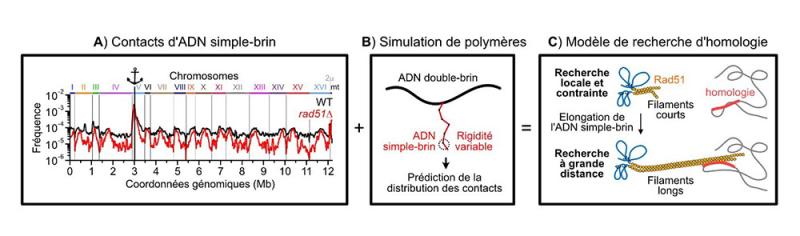
Figure :
(A) La technique du ssHi-C permet de déterminer la fréquence de contact entre l’ADN simple-brin généré au niveau d’une cassure (représenté par l’ancre) et le reste du génome dans un contexte sauvage ou mutant. (B) Ces données expérimentales sont comparées aux prédictions de simulations de polymères, (C) ce qui conduit à un modèle d’expansion de la recherche d’homologie basée sur la formation de long filaments rigides formés par la protéine Rad51 de part et d’autre de la cassure.
Pour en savoir plus :
Mechanism of homology search expansion during recombinational DNA break repair in Saccharomyces cerevisiae.
A. Dumont, N. Mendiboure, J. Savocco, L. Anani, P. Moreau, A. Thierry, L. Modolo, D. Jost, A. Piazza
Molecular Cell, 22 août 2024. DOI : 10.1016/j.molcel.2024.08.003
Contact
Laboratoire
Laboratoire de Biologie et Modélisation de la Cellule - LBMC ( CNRS / ENS de Lyon)
Ecole Normale Supérieure de Lyon
46 Allée d’Italie
69007 Lyon
