Du nouveau sur la régulation des mécanismes de réparation de l’ADN
Lorsque l’ADN est cassé, une protéine en forme de perle et appelée Ku s’enfile à partir des extrémités de la cassure. Dans un article publié dans Cell reports, les scientifiques décrivent les mécanismes qui permettent à la cellule d’empêcher une accumulation excessive de Ku sur l’ADN. Ces mécanismes, parfois déficients dans les cellules tumorales, protègent l’expression du génome lors de la réparation de l’ADN.
De nombreuses thérapies anticancéreuses, comme la radiothérapie, assurent leur efficacité en induisant des cassures double-brin de l’ADN, c’est-à-dire la rupture des deux brins de la molécule d’ADN, générant ainsi deux extrémités. Chez l’homme, le complexe protéique Ku se lie immédiatement sur chaque extrémité des cassures grâce à sa structure en anneau. Cet anneau à la capacité de glisser sur l’ADN et de multiples complexes Ku peuvent ainsi se charger à partir de l’extrémité, puis glisser à leur tour comme des perles sur un collier. Or, l’ADN couvert par Ku ne peut plus s’ouvrir pour être copié ou pour permettre l’expression des gènes. On suspectait donc l’existence de mécanismes bloquant ce glissement dans les cellules.
Une protéine retient Ku à l’extrémité des cassures.
Dans un article publié dans Cell Reports, des scientifiques ont utilisé la microscopie de fluorescence à super-résolution, une technique permettant notamment d’étudier la composition des complexes de réparation au niveau de chaque cassure pour suivre la quantité de Ku se chargeant sur des cassures induites dans des cellules humaines par les rayons-X, mimant la radiothérapie. Avec cette approche les scientifiques ont découvert que la protéine DNA-PKcs, qui se lie directement à Ku à l’extrémité des cassures, bloque son glissement sur l’ADN. Bien que la DNA-PKcs soit connue pour son activité kinase, qui lui permet de moduler par phosphorylation les propriétés de plusieurs protéines de réparation, les scientifiques ont montré que, dans ce cas, il s’agissait d’un rôle physique de la DNA-PKcs. Ils ont également montré que ce rôle était retrouvé chez le xénope, un organisme modèle que les chercheurs ont utilisé pour des analyses fines de la composition des complexes de réparation.
Deux mécanismes limitent l’accumulation de Ku dans la chromatine.
De plus, les ils ont montré que lorsque la DNA-PKcs est absente, deux mécanismes limitent l’accumulation de Ku sur l’ADN. Ku peut être ainsi modifié par greffage de multiples copies d’une protéine appelée ubiquitine, un processus appelé ubiquitination. La chaine de poly-ubiquitine résultante constitue alors un signal pour son extraction de l’ADN. Ce premier mécanisme dépend de la protéine FBXL12 qui recrute un complexe de greffage de l’ubiquitine. Par ailleurs, lorsque les cassures sont induites pendant la phase de duplication de l’ADN, l’excès de Ku aux cassures est éliminé par digestion de l’ADN par un complexe nucléase régulé par la protéine CtIP et par la kinase ATM.
L’excès de Ku bloque l’expression des gènes proches.
Enfin, les scientifiques ont étudié l’impact que pourrait avoir un glissement excessif de Ku sur l’expression des gènes, qui nécessite l’ouverture de la double-hélice d’ADN. Ils ont pour cela développé un outil mesurant l’expression d’un gène à proximité d’extrémités d’ADN. Ils ont ainsi pu confirmer qu’en absence de la DNA-PKcs, l’expression des gènes voisins est bloquée par l’accumulation de Ku sur l’ADN.
Ces résultats apportent une réponse à une question fondamentale qui restait jusqu’alors en suspens : comment la quantité de Ku se chargeant sur les cassures de l’ADN est-elle régulée ? Ils identifient également des cibles thérapeutiques potentiellement utilisables pour tuer des cellules tumorales porteuses de défauts spécifiques de certains de ces mécanismes.
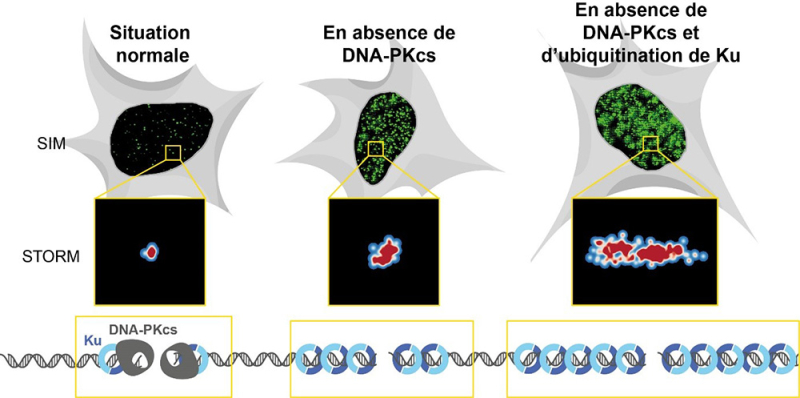
Figure : En situation normale, la DNA-PKcs s’associe à Ku à l’extrémité des cassures et bloque son entrée sur l’ADN. En absence de DNA-PKcs, Ku s’accumule sur l’ADN, mais cette accumulation est limitée par une élimination active dépendante de sa modification par des chaines d’ubiquitine. Lorsque ces deux systèmes sont absents, Ku s’accumule de façon excessive. L’accumulation de Ku au niveau de cassures individuelles peut être quantifiée par imagerie de fluorescence en mesurant l’intensité des foyers de Ku (images en SIM) ou en suivant la taille des foyers de Ku par microscopie à super-résolution de type STORM.
En savoir plus : Identification of the main barriers to Ku accumulation in chromatin. Madeleine Bossaert, Andrew Moreno, Antonio Peixoto, Marie-Jeanne Pillaire, Pauline Chanut, Philippe Frit, Patrick Calsou*, Joseph John Loparo*, Sébastien Britton*. Cell Rep (2024). DOI:https://doi.org/10.1016/j.celrep.2024.114538
Contact
Laboratoire
Institut de pharmacologie et biologie structurale - IPBS (CNRS/Université de Toulouse III - Paul Sabatier)
205 route de Narbonne
31077 Toulouse
