La régulation de la dégradation des ARN messagers est indispensable au développement des plantes
Les ARNm, ou ARN messagers, sont des copies transitoires d’un gène utilisées comme intermédiaires pour la synthèse des protéines. Ces molécules sont sujettes à un grand nombre de régulation qui affecte notamment leur stabilité. Dans une étude publiée dans la revue Nucleic Acids Research, les scientifiques ont mis en évidence l’importance de la dégradation des ARNm pour le bon développement des organes chez les plantes.
La dégradation des ARNm est un processus évolutivement conservé qui permet de réguler leur stabilité. Cette dégradation peut se mettre en place par différentes voies enzymatiques. L’importance relative de ces différentes voies de dégradation n’a jamais été clairement élucidée à ce jour. Dans un article publié récemment dans la revue Nucleic Acids Research, les scientifiques ont réussi à élucider l’importance d’une de ces voies de dégradation chez la plante modèle Arabidopsis thaliana. L’enzyme XRN4 est impliquée dans deux voies de dégradation des ARNm, l’une, appelée dégradation 5’-3’ cytosolique qui se produit dans le cytoplasme de la cellule et l’autre appelée dégradation 5’-3’ co-traductionnelle qui se déroule au niveau du ribosome lorsque le messager est traduit par ce dernier. En se basant sur des données de prédiction de structure, ils ont réussi à inactiver spécifiquement la dégradation 5’-3’ co-traductionnelle tout en maintenant une dégradation 5’-3’ cytosolique active. Par des approches de séquençage massif, les scientifiques ont ainsi pu déterminer l’importance de la dégradation 5’-3’ co-traductionnelle dans la stabilité des ARN messagers. Au niveau des feuilles, l’absence de dégradation 5’-3’ co-traductionnelle provoque une stabilisation massive de plusieurs milliers de molécules d’ARNm démontrant l’importance cruciale de cette voie dans la stabilité des ARN messagers. En revanche, au niveau des racines, l’absence de cette voie de dégradation provoque une déstabilisation encore plus importante de milliers de molécules d’ARNm. Les scientifiques ont montré qu’un mécanisme de compensation se mettait alors en place pour pallier l’absence de dégradation 5’-3’ co-traductionnelle au niveau des racines.
Enfin, l’absence de cette dégradation au sein de la plante provoque des défauts morphologiques au niveau des feuilles et des racines laissant supposer l’importance cruciale de la dégradation des ARNm pour le bon développement des organes chez les plantes.
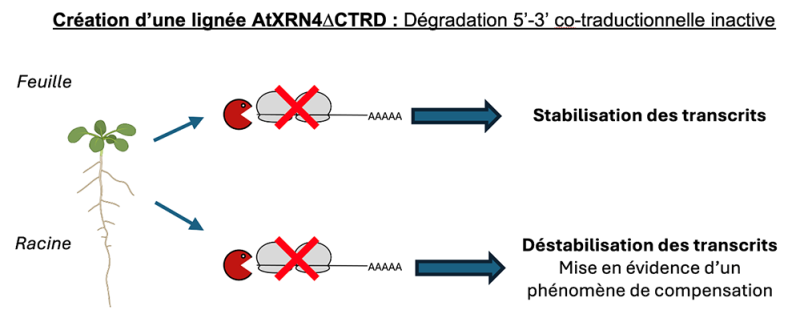
Figure : En créant une lignée transgénique spécifiquement impactée dans la dégradation co-traductionnelle (lignée AtXRN4∆CTRD), il a été alors possible de démontrer que l’absence de cette voie de dégradation induit une stabilisation de plusieurs milliers de transcrits dans les feuilles d’Arabidopsis. En revanche au niveau des racines, son absence induit une déstabilisation de plusieurs milliers de transcrits mettant en évidence un mécanisme de compensation pour maintenir une dégradation active au sein de cet organe.
En savoir plus : Genome-wide analysis of mRNA decay in Arabidopsis shoot and root reveals the importance of co-translational mRNA decay in the general mRNA turnover. M.C. Carpentier, A.E Receveur, A. Boubegtitene, A. Cadoudal, C. Bousquet-Antonelli, and R. Merret. Nucleic Acids Research, 9 mai 2024.
Contact
Laboratoire
Laboratoire génome et développement des plantes (CNRS/Université de Perpignan)
58 Avenue Paul Alduy
66000 Perpignan