Comment un ADN mobile trouve sa cible dans le génome, révélé au niveau atomique
Les scientifiques, dans une étude publiée dans la revue Nature communications ont identifié le mécanisme permettant à certains rétrotransposons, également connus sous le nom d’ADN mobiles, de s'insérer dans des régions du génome pauvres en gènes où leur insertion est moins délétère. Ces résultats pourront être utiles pour l'amélioration des vecteurs viraux utilisés en thérapie génique afin de soigner des maladies génétiques et des cancers.
Les éléments transposables sont des séquences d’ADN mobiles capables de se déplacer dans les génomes et de les modifier suite à leur insertion. Ils contribuent grandement à la plasticité des génomes eucaryotes, façonnant l'évolution et l'adaptation des espèces, mais participent également à l’émergence de maladies génétiques ou des cancers.
La localisation des éléments transposables n’est pas aléatoire au sein des génomes, révélant leur accumulation dans des régions spécifiques. Ceci résulte en partie de processus de sélection naturelle qui éliminent les nouvelles insertions fortement délétères et maintiennent les insertions bénéfiques. Mais pour certains éléments transposables, elle est aussi le fruit d’un processus actif qui cible leur intégration dans des régions spécifiques du génome, où les insertions auront peu ou pas d'effet néfaste sur la physiologie de l'organisme. C’est le cas du rétrotransposon Ty1 de la levure de bière Saccharomyces cerevisiae : il s’insère dans une région étroite à l’échelle du génome, située en amont des gènes transcrits par l'ARN polymérase III, un complexe enzymatique qui se lie à l'ADN pour synthétiser l'ARN. La protéine intégrase codée par Ty1 catalyse son insertion dans le génome de la levure.
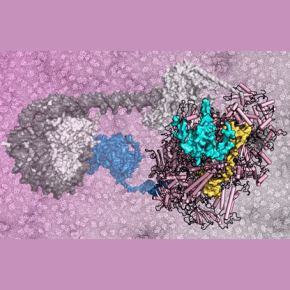
En utilisant la cryo-microscopie électronique et en validant leurs observations par des approches de génétiques moléculaires, les scientifiques fournissent des détails à l’échelle atomique de l'interaction entre l'ARN polymérase III et la protéine intégrase. Ces travaux décrivent notamment comment une région dépliée de la protéine intégrase se positionne dans une fissure à la surface de l'ARN polymérase III. Cette interaction reconfigure l'ARN polymérase III de manière à ce qu'elle soit piégée plus longtemps sur l'ADN, augmentant ainsi les chances d'insérer le rétrotransposon dans des endroits sûrs du génome dépourvus de gènes essentiels. Un tel mécanisme de reconfiguration d’une activité cellulaire au bénéfice de l’insertion d’un rétrotransposon est remarquable et n’avait encore jamais été démontré à ce jour.
En plus de constituer une avancée majeure pour la recherche fondamentale, cette découverte pourrait contribuer à l’amélioration des vecteurs de transfert de gènes utilisés en thérapie génique pour introduire un gène correcteur dans les chromosomes. En effet, un des problèmes majeurs à surmonter en thérapie génique est d’insérer un gène sans endommager les gènes de l’organisme, c’est à dire soigner une maladie sans en déclencher une autre. Ces nouvelles informations atomiques sur l'interaction entre l’intégrase de Ty1 et l’ARN polymérase III, pourront être utilisées pour concevoir une nouvelle génération de vecteurs de thérapie génique qui cibleront majoritairement des régions sûres du génome humain, ce qui devrait réduire leur potentiel mutagène.

Figure : En haut : le rétrotransposon Ty1 s’insère en amont des gènes transcrits par l’ARN polymérase III (Pol III), grâce à une interaction entre son intégrase (IN1) et la protéine AC40 pour la Pol III. En bas : L’analyse par cryo-microscopie électronique des complexes formées entre l’intégrase et la Pol III (à droite) a révélé les détails au niveau atomique de cette interaction (encart à gauche).
En savoir plus :
Structural basis of Ty1 integrase tethering to RNA polymerase III for targeted retrotransposon integration.
Nguyen, P.Q., Huecas, S., Asif-Laidin, A. et al.
Nature Communications, 28 March 2023. DOI: https://doi.org/10.1038/s41467-023-37109-4
Contact
Laboratoires
Institut de recherche Saint-Louis – IRSL (CNRS/Inserm/Université Paris Cité)
Unité Génomes, biologie cellulaire et thérapeutique
Équipe Biologie des génomes : de l’ADN mobile à la dynamique des chromosomes
Bâtiment Jean Bernard
1 avenue Claude Vellefaux
75010 Paris
Group Structure of Macromolecular Assemblies
Centro de Investigaciones Biológicas Margarita Salas,
Consejo Superior de Investigaciones Científicas (CSIC)
28040 Madrid