Des "pièges à ribosome" pour éteindre la traduction des ARN messagers hox
Dans cet article, publié dans la revue eLife, les scientifiques ont "décortiqué" le mécanisme moléculaire de la régulation de la traduction des ARN messagers des gènes hox. Ces gènes sont exprimés de manière séquentielle au cours des stades précoces du développement chez les vertébrés. A tour de rôle, ils sont allumés puis éteints à des étapes clés du développement. Les chercheurs ont découverts dans les extrémités 5’ non traduites, des éléments régulateurs qui piègent les ribosomes. Ces éléments sont essentiels pour éteindre efficacement l’expression des gènes hox aux moments opportuns.
Les ARN messagers (ARNm) transcrits à partir des gènes hox permettent la synthèse d’une famille de protéines appelées homéoprotéines. Ces protéines sont conservées chez tous les vertébrés. Elles participent notamment à l’élaboration précise de la colonne vertébrale pendant les phases très précoces du développement de l’embryon. Pour ce faire, les ARNm hox, qui sont présents en nombre dans l’embryon, sont "allumés" et puis "éteints" de manière séquentielle selon un programme clairement établi. La précision de cet enchainement est primordiale pour garantir un développement harmonieux de l’embryon. Ceci implique que les ARNm hox doivent être non seulement allumés à des étapes clés du développement mais également éteints à d’autres moments. Le mécanisme d’allumage de ces ARN messagers hox a été décrypté récemment. En revanche, on ignorait comment la traduction en proteines de ces ARNm est réprimée lorsque le programme développemental l’exige.
Les travaux réalisés par les chercheurs ont permis de comprendre comment ces ARN messagers sont maintenus non traduits lorsque le programme développemental l’exige. Ils contiennent dans leur extrémité 5’ non traduites (5’UTR), des éléments structuraux qui piègent le ribosome et en dissocient les sous-unités, l’empêchant d’atteindre la séquence codante des protéines hox, qui ne pourra donc pas être traduite. De tels éléments, qui contiennent des éléments de structures secondaires et des petites phases de lecture, également appelés uORF (usptream Open Reading Frame), ont été cartographiés et décryptés dans le cas des ARN messagers hoxa3 et hoxa11. Ces travaux permettent de mieux comprendre le ballet moléculaire mis en œuvre pendant les étapes précoces du développement de l’embryon.
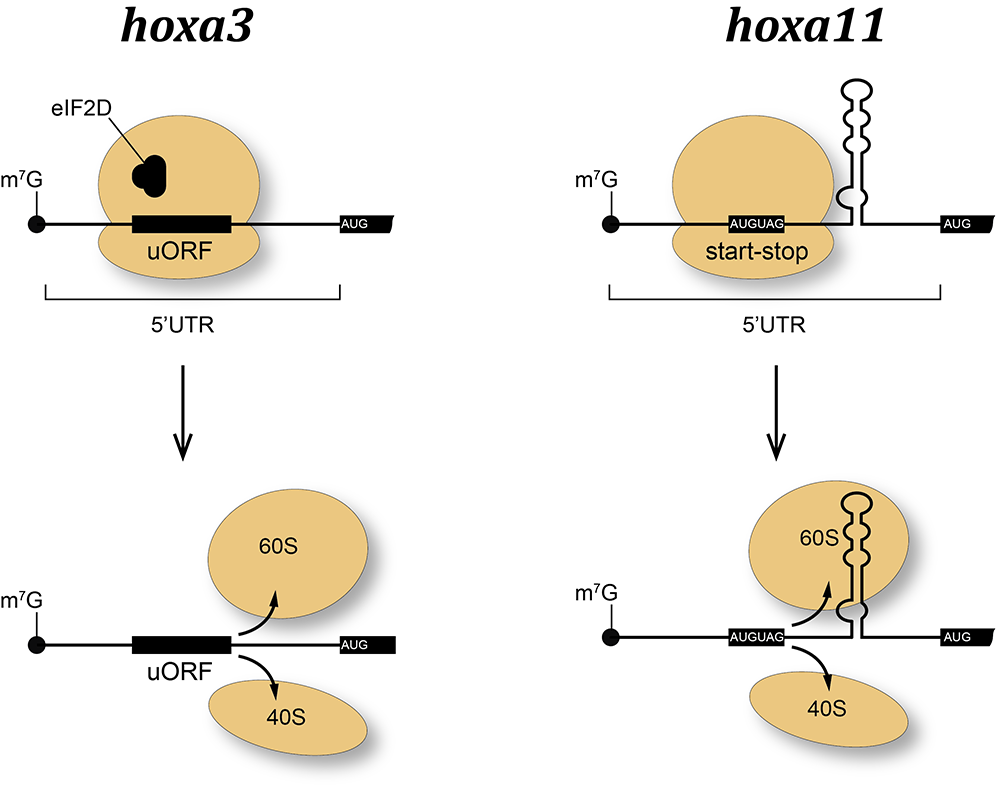
Figure: modèle de la régulation de la traduction des ARN messagers hoxa3 et hoxa11.
Les ARN messagers hoxa3 et hoxa11 sont régulés grâce à deux mécanismes différents. L’extrémité 5’ non traduite (5’UTR) de l’ARN messagers hoxa3 contient une petite phase de lecture appelée uORF (‘upstream Open Reading Frame’) qui piège le ribosome. A la fin de l’uORF, le ribosome est dissocié et ne peut atteindre la phase codante principale de la protéine hoxa3. Ce mécanisme requiert un facteur spécifique appelé eIF2D. La région 5’UTR de l’ARN messager hoxa11 contient deux éléments qui permettent également de piéger le ribosome, un doublet de codons ‘start-stop’ et une grande structure en épingle à cheveux. Ces deux éléments vont également piéger le ribosome qui va ensuite se dissocier avant d’atteindre la phase principale de la protéine hoxa11.
Pour en savoir plus :
Translation inhibitory elements from Hoxa3 and Hoxa11 mRNAs use uORFs for translation inhibition.
Alghoul F, Laure S, Eriani G, Martin F.
Elife. 2021 Jun 2;10:e66369. doi: 10.7554/eLife.66369.
Contact
laboratoire
Institut de biologie moléculaire et cellulaire
2 allée Konrad Roentgen
67084 Strasbourg cedex