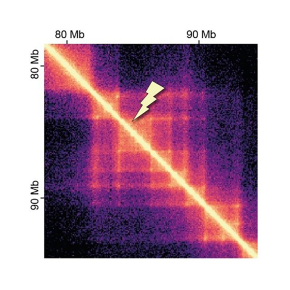
Les boucles d’ADN au service de la réparation du génome.
Suite à l’apparition d’une cassure double-brin dans l’ADN, la chromatine qui entoure ce dommage est modifiée sur un grand domaine de l’ordre d’un million de paires de bases, constituant ainsi un foyer de réparation qui permet la prise en charge de la cassure. En étudiant l’organisation spatiale de la chromatine par des techniques de Capture de Conformation des Chromosomes, les scientifiques ont montré que les domaines génomiques riches en interactions appelés TAD sont des unités fonctionnelles de la réponse aux cassures de l’ADN. Ces travaux, publiés dans la revue Nature, mettent en évidence que le processus d’établissement des TAD par extrusion de boucle joue un rôle majeur dans la formation rapide de ces foyers favorables à la réparation de l’ADN.
Les cassures double-brin de l’ADN (DSB pour Double-Strand Break) sont des dommages extrêmement toxiques et leur réparation est donc essentielle pour préserver l’intégrité des chromosomes. En réponse à l’apparition d’une DSB dans le génome, la kinase ATM permet en quelques minutes la phosphorylation du variant d’histone H2AX sur un domaine chromatinien de très grande taille, de l’ordre d’une mégabase (un million de paires de bases), qui constituera ainsi un foyer de réparation. Néanmoins, la façon dont ces foyers sont assemblés aussi rapidement pour établir un environnement nucléaire qui soit favorable à la réparation des cassures de l’ADN restait encore largement méconnue.
Le génome est organisé en trois dimensions dans le noyau des cellules et l'ADN, en association avec des protéines (les histones) forme une structure complexe appelée chromatine. Les TAD (pour Topologically Associating Domains) sont des domaines enrichis en interactions chromatiniennes qui correspondent chacun à une unité clé de cette organisation de la chromatine. Il a été proposé que les TAD émergent d’un processus d’"extrusion de boucle". Selon ce modèle, une fois associé à la chromatine, l’anneau de cohésines entoure l’ADN pour former une boucle, qui s’élargit progressivement à mesure que les cohésines extrudent activement l’ADN. Ce processus prend fin lorsque les cohésines rencontrent des séquences particulières appelées "éléments frontières forts" qui marquent les limites du TAD. Ces boucles d’ADN sont omniprésentes sur le génome humain et différentes études ont mis en lumière un rôle fonctionnel de l’organisation de l’ADN en TAD dans des processus tels que la transcription ou la réplication. Toutefois, le rôle des TAD et de leur mécanisme de formation par extrusion de boucle n’avait pas encore été impliqué dans la réparation de l’ADN.
Pour répondre à ces interrogations, les chercheurs ont utilisé des techniques de capture de conformation des chromosomes qui permettent de connaitre respectivement les interactions chromatiniennes du génome entier ou d’un locus d’ADN spécifique avec le reste du génome. Ces technologies, combinées à un système cellulaire dans lequel il est possible d’induire un nombre constant de DSB à des positions connues et annotées du génome (modèle DIvA), ont permis de mettre en évidence un rôle majeur de la conformation de la chromatine par extrusion de boucle dans la réparation des DSB. En effet, les chercheurs ont montré que la phosphorylation de l'histone H2AX sur la longueur d'un TAD entier est permise grâce à un processus d’extrusion de boucle de chromatine dépendant des cohésines et ayant lieu de part et d’autre de chaque DSB. Les auteurs proposent ainsi un nouveau modèle permettant d’expliquer d’une part, comment sont assemblés les foyers de réparation, et d’autre part, la rapidité de cet assemblage, et impliquant les TAD comme unités fonctionnelles de la réponse cellulaire aux dommages à l’ADN.
Ces travaux ont permis de montrer le rôle majeur de la conformation des chromosomes dans la maintenance de l’intégrité du génome tout en mettant en évidence pour la première fois un exemple de modification de la chromatine grâce au processus d’extrusion de boucle.
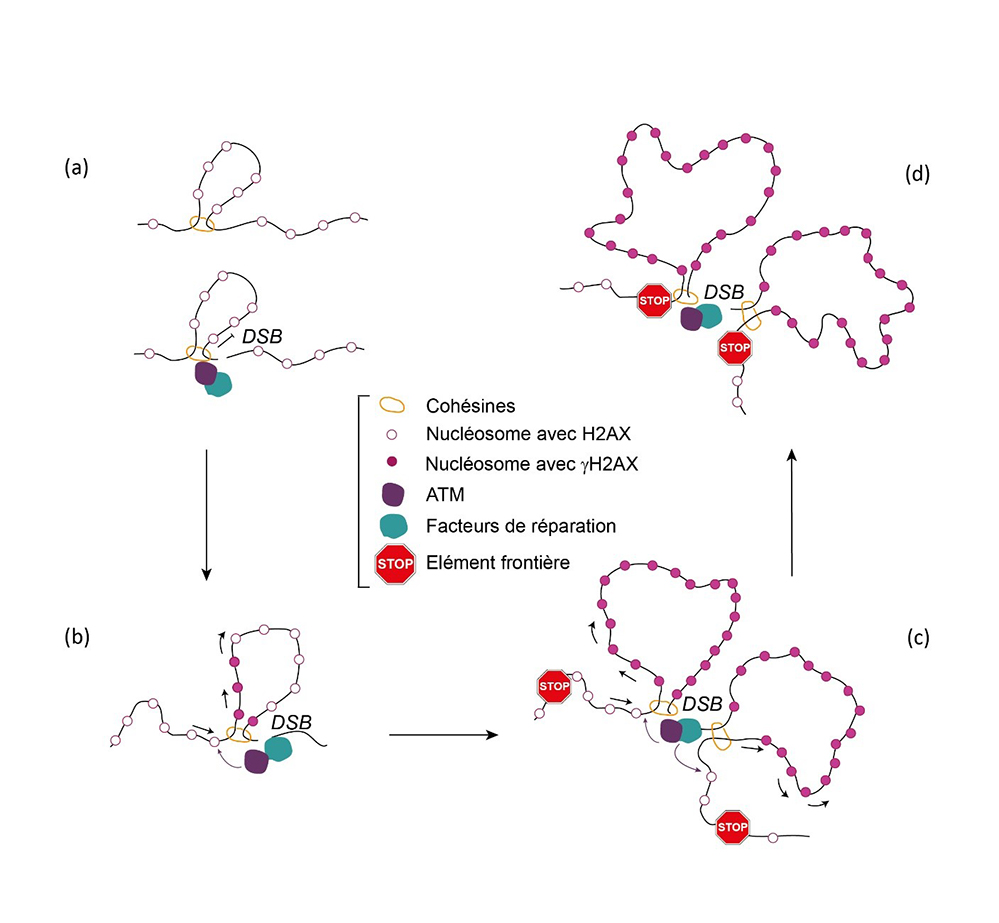
Figure : Le processus d’extrusion de boucle de chromatine médié par les cohésines aux DSB assure l’établissement des foyers de réparation.
(a) Les phénomènes d’extrusion de boucle réalisés par les cohésines se produisent en continu sur le génome. L’apparition d’une cassure double-brin dans l’ADN (DSB) provoque un blocage de ces processus en cours, conduisant à une accumulation des cohésines aux DSB. (b) Les cohésines, bloquées d’un côté par la DSB, assurent un processus d’extrusion de boucle unidirectionnel et ancré à la DSB. La kinase ATM, qui n’est recrutée que localement aux DSB, phosphoryle H2AX pour former gamma-H2AX à mesure que les nucléosomes sont extrudés. (c) Le même processus se produit de chaque côté des DSB, permettant des phénomènes d’extrusion de boucle unidirectionnels et divergents, et donc un établissement bidirectionnel de gamma-H2AX. (d) Ces processus d’extrusion de boucle s’arrêtent lorsque les cohésines rencontrent des éléments frontières forts. Puisque la vitesse d’extrusion de boucle de chromatine mesurée in vitro peut atteindre 0,5-2 kb/sec, ce mécanisme pourrait permettre de phosphoryler H2AX sur un TAD entier (~ 1-2 Mb) en 10-30 min, permettant la mise en place rapide des foyers de réparation.
Pour en savoir plus :
Loop extrusion as a mechanism for formation of DNA damage repair foci.
Arnould C, Rocher V, Finoux AL, Clouaire T, Li K, Zhou F, Caron P, Mangeot PE, Ricci EP, Mourad R, Haber JE, Noordermeer D, Legube G.
Nature. 2021 Feb 17. doi: 10.1038/s41586-021-03193-z. Online ahead of print.
Contact
Laboratoire
Centre de Biologie Intégrative (CBI) de Toulouse (CNRS/Université Paul Sabatier)
169 Avenue Marianne Grunberg-Manago
31062 Toulouse Cedex 9