Visualiser les mouvements des protéines intrinsèquement désordonnées en temps réel
Dans cette étude pluridisciplinaire, les scientifiques décrivent une méthode très puissante qui permet de visualiser les mouvements des "protéines intrinsèquement désordonnées" à l’échelle de la molécule unique. Ces résultats, qui ouvrent de nouvelles perspectives pour la caractérisation de ces protéines dont l’étude est très difficile sont publiés dans la revue Nature Nanotechnology.
Les protéines intrinsèquement désordonnées (« Intrinsically Disordered Proteins », IDPs) sont très répandues dans le monde vivant et caractérisées par leur incapacité à adopter une structure tridimensionnelle unique et précise. Ainsi, leur forme (ou conformation) change en permanence. Elles peuvent être assimilées à des spaghetti en cours de cuisson remuant sans cesse dans une casserole d’eau bouillante. Cependant elles restent capables d’exercer une fonction biologique qui repose précisément sur cette absence de structure fixe. Leur malléabilité leur permet d’établir des interactions avec de nombreux partenaires. De ce fait, les IDPs sont souvent impliquées dans des phénomènes de régulation de processus biologiques. Leur dysfonctionnement peut conduire à de nombreuses pathologies telles que le cancer, les maladies cardiovasculaires et les maladies neurodégénératives. Lors de l’interaction avec leurs partenaires, les IDPs peuvent partiellement se replier, c'est à dire subir une transition désordre-ordre, et adopter une structure rigide sur au moins une partie de leur chaine d'acides aminés. Ces régions ont souvent une propension à se replier partiellement même en l’absence de partenaire. Identifier de telles régions est important car il s’agit très souvent de sites impliqués dans la reconnaissance de partenaires. Ces régions constituent donc des cibles potentielles dans des approches thérapeutiques. De plus, il est important de quantifier la tendance de ces régions à se replier car cela préfigure la propension de l’IDP à se lier à son partenaire.
Pour toutes ces raisons, il est important de pouvoir obtenir des informations structurales sur ces protéines. Le fait que leur forme change pose toutefois un défi majeur dans la quête de cette information. Les techniques les plus utilisées pour l’étude des IDPs fournissent des informations qui sont "moyennées" sur l’ensemble des formes que l’IDP peut adopter/explorer, et sont incapables d’examiner les différentes conformations individuelles. Si des approches alternatives permettant de contourner cette limitation existent, elles souffrent néanmoins d’autres limitations techniques. Ainsi, il existe un réel besoin de techniques adaptées à l’étude structurale des IDPs.
La microscopie à force atomique (« Atomic Force Microscopy », AFM) a la capacité unique de visualiser à haute résolution des échantillons biologiques déposés sur des surfaces inertes (typiquement du mica). La visualisation repose sur la capacité d’une pointe de dimensions atomiques à explorer la forme de la macromolécule biologique (une protéine ou un acide nucléique notamment, ou encore un complexe entre les deux ..) déposée sur la surface inerte. Les mouvements de cette pointe sont en suite transformés en images. Les processus dynamiques impliquant les biomolécules se produisant sur une échelle de temps très courte (milliseconde voire moins), il est important, pour pouvoir les décrire, de disposer d’une technologie à haute vitesse, permettant d’enregistrer une image en un lapse de temps très court (de l’ordre de 30 à 60 millisecondes). Or, c’est précisent ce que fait la microscopie à force atomique a grande vitesse (« High Speed Atomic Force Microscopy », HS-AFM). Ainsi il devient possible de visualiser de manière directe les changements structuraux et les interactions se produisant au cours du temps dans les macromolécules biologiques individuelles, ce qui n'est actuellement pas possible avec d'autres techniques.
Dans le cadre d'une collaboration avec l’Université de Kanazawa (Japon), les chercheurs ont utilisé la HS-AFM pour étudier plusieurs IDPs, et ont obtenu des films permettant de visualiser leurs mouvements à l’échelle de la molécule unique. A partir de ces films, en faisant un « arrêt sur image » et en étudiant un grand nombre de ces images, il est possible d’extraire toute une série de paramètres physiques (dimensions, épaisseur, présence ou absence de globules). On peut ainsi identifier les régions qui restent désordonnées en permanence et celles qui, au contraire, se replient, et cela avec une précision approchant le niveau de l’acide aminé. On peut également quantifier la propension de ces régions à se replier. Enfin, l’HS-AFM fournit des informations sur la nature structurale des régions repliées de manière transitoire, en permettant notamment de discriminer entre une hélice α et un repliement non régulier.
L’HS-AFM est donc une technique très puissante pour caractériser les IDPs. En permettant de visualiser et analyser des molécules uniques elle permet de résoudre leur hétérogénéité conformationnelle et ainsi d'en apprendre beaucoup plus que dans l'analyse d’une population de molécules dont on ne peut extraire que des valeurs moyennes.
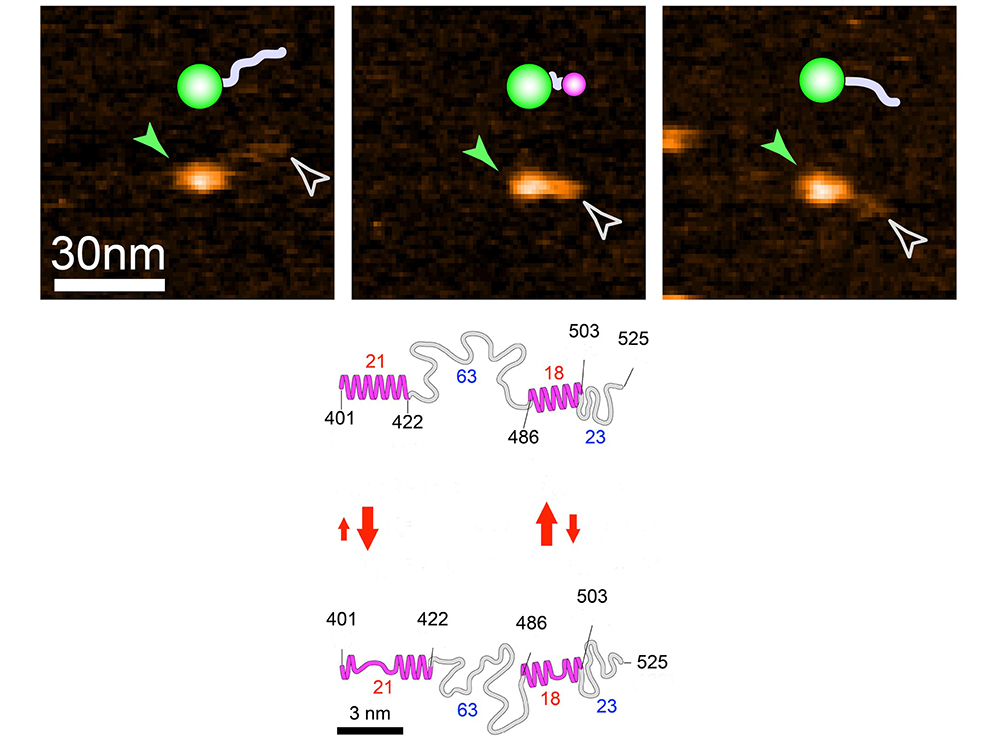
Figure :
Haut : Images HS-AFM et schémas associés de la protéine de fusion NTAIL-GFP montrée dans trois états différents. NTAIL, ici représentée en gris, est une IDP de 125 résidus, correspondant au domaine C-terminal de la nucléoprotéine du virus de la rougeole. En vert est représentée la Green Fluorescent Protein (GFP), ici utilisée comme « boulet » pour « alourdir » NTAIL et en empêcher les mouvements autrement trop rapides pour pouvoir être visualisés. En rose est représenté le petit globule décelable à l’extrémité N-terminale de la protéine de fusion. La pointe de flèche verte indique le globule plus grand (correspondant à la GFP), alors que la pointe de flèche blanche indique l’extrémité de la région désordonnée.
Bas : Schéma montrant les caractéristiques structurales et dynamiques de NTAIL. Le schéma supérieur et inférieur correspondent à l’état le plus et le moins ordonné (replié), respectivement. Les nombres en rouge et bleu représentent les nombres de résidus contenus dans les petits globules et les régions complètement désordonnées, respectivement. Les flèches rouges indiquent la nature cinétique des changements de la hauteur du globule, et leur dimension indique la direction vers laquelle l’équilibre est déplacé. Les nombres en noir correspondent aux positions des acides aminés (NTAIL est numérotée de 401 à 525).
Les molécules de NTAIL-GFP obtenu par HS-AFM
Film obtenu par HS-AFM (High Speed Atomic Force Microscopy) de NTAIL-GFP. Les molécules de NTAIL-GFP ont été placées sur une surface de mica dans un tampon phosphate 10 mM (pH 6,0) et filmées à 10 fps pour une zone de balayage de 80 x 100 nm2 avec 80 x 80 pixels. Ce film montre les mouvements en temps réel. © Sonia Longhi, Noriyuki Kodera & Toshio Ando
Audiodescription
Pour en savoir plus
Structural and dynamics analysis of intrinsically disordered proteins by high-speed atomic force microscopy
Kodera N, Noshiro D, DoraSK , Mori T, Habchi J, Blocquel D, Gruet A, Dosnon M, Salladini E, Bignon C, Fujioka Y, Oda T, Noda NN, Sato M, Lotti M, Mizuguchi M, Longhi S and Ando T
Nature Nanotechnology 23 Nov 2020. https://doi.org/10.1038/s41565-020-00798-9
Les équipes :
Contact
Laboratoires
Architecture et Fonction des macromolécules Biologiques (AFMB) - (CNRS/Aix-Marseille Université)
163, Avenue de Luminy, Case 932, 13288 Marseille Cedex 09
Nano Life Science Institute (WPI NanoLSI)
Kanazawa University
Kakuma-machi
Kanazawa 920-1192
