Le repliement tridimensionnel des chromosomes en super-résolution
En combinant le marquage fluorescent d’ADN à la microscopie en super-résolution, les scientifiques ont caractérisé la structure de domaines génomiques appelés TADs au sein desquels les interactions chromatiniennes sont enrichies. Cette organisation dépend du complexe cohésine, qui génère ces interactions, et de la protéine CTCF, qui prévient les contacts entre TADs voisins. Les TADs sont en outre subdivisés en « nanodomaines » qui dépendent du profil épigénétique de la chromatine. Ces résultats sont publiés dans la revue Nature Genetics.
L’étude des mécanismes de l’organisation tridimensionnelle du génome révèle des relations fonctionnelles entre la structure des chromosomes et la régulation génique. Au sein du noyau des cellules eucaryotes, le génome apparaît replié de manière hiérarchique. A la plus petite échelle, la molécule d’ADN s’enroule autour des protéines histones, formant les nucléosomes et la structure primaire de la fibre de chromatine. A plus grande échelle, chaque chromosome occupe son propre territoire au sein du noyau. Entre ces deux extrêmes, l’organisation physique des chromosomes reste peu caractérisée, alors que c’est à cette échelle que la plupart des processus fonctionnels, comme la réplication de l'ADN et la transcription en ARN se produisent. Durant la dernière décennie, l’utilisation d’une méthode de biologie moléculaire sophistiquée appelée Hi-C, qui permet la cartographie de l’ensemble des contacts chromosomiques, a révélé la présence de domaines génomiques de l’ordre de la Mégabase (un million de paires de bases) enrichis en interactions : les « Topologically Associating Domains » ou TADs. Différentes études ont mis en lumière un rôle fonctionnel des TADs dans la régulation génique, notamment par leur capacité à contenir les contacts à longue distance entre les promoteurs (sites de régulation de la transcription) des gènes et des séquences régulatrices telles que les « enhancers ». La perturbation de ces domaines est ainsi associée à des maladies développementales ou à certains cancers. Cependant, la méthode de Hi-C générant des cartes d’interactions chromosomiques obtenues à partir de larges populations de cellules, une caractérisation directe de la structure de ces domaines au niveau de la cellule isolée manquait pour comprendre comment le génome est physiquement replié en TADs.
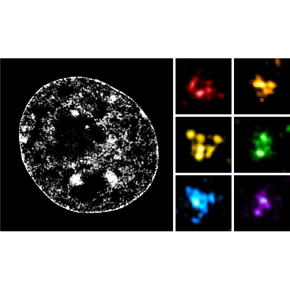
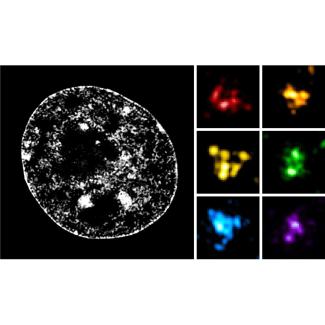
Dans cette étude, les chercheurs ont combiné une technique de marquage spécifique de l’ADN utilisant des oligonucléotides fluorescents appelée « Oligopaint Fluorescent in situ hybridization » avec de la microscopie en super-résolution pour étudier la structure des TADs avec une résolution spatiale de l’ordre de la centaine de nanomètres (milliardièmes de mètre). L’analyse de la structure de nombreuses régions chromosomiques dans des cellules souches embryonnaires de souris a révélé une grande hétérogénéité structurale des TADs. Malgré cette variabilité, la majorité des cellules présente des contacts enrichis à l’intérieur d'un TAD donné par rapport aux TADs voisins. Ces résultats sont donc consistants avec le rôle proposé des TADs dans la définition physique de domaines génomiques fonctionnels. De plus, leur ségrégation spatiale est exacerbée dans les cellules progénitrices neurales par rapport aux cellules souches, ce qui suggère que le repliement des chromosomes en TADs s’intensifie lors de la différentiation cellulaire.
Les chercheurs ont également étudié les effets des déplétions de la protéine CTCF, localisée au niveau des frontières entre TADs, et de la sous-unité RAD21 du complexe cohésine, toutes deux nécessaires à la formation des TADs. En l’absence de l’une ou l’autre de ces protéines, les contacts chromatiniens préférentiels au sein des TADs sont abolis, confirmant les précédentes observations de Hi-C. Les résultats révèlent, de plus, que la disparition des TADs s’effectue selon deux mécanismes distincts. La déplétion de CTCF entraîne des interactions anarchiques entre TADs voisins, mettant en évidence son rôle de protéine barrière. La déplétion de RAD21 entraîne en revanche une perte de contacts à l’intérieur des TADs, révélant son rôle dans la génération d’interactions chromatiniennes. Ces résultats, consistants avec un modèle de formation de TADs par extrusion de boucles, indiquent que le complexe cohésine est responsable de l’enrichissement de contacts chromatiniens, tandis que CTCF, en définissant les frontières, contraint l’action de la cohésine au sein des TADs.
Enfin, l’utilisation de la microscopie en super-résolution a révélé la présence de sous-structures à l’intérieur des TADs, appelées « nano-domaines chromatiniens » ou CNDs. Leur organisation dépend de l’état épigénétique de la chromatine et de l’hyper-acétylation des histones qui, en antagonisant les interactions entre nucléosomes, perturbe la formation des CNDs. Ces données suggèrent donc que les CNDs se forment par agrégation de nucléosomes.
Cette étude caractérise ainsi l’organisation physique des TADs en cellules uniques, clarifie les rôles de CTCF et cohésine dans leur formation, et révèle l’existence de CNDs, qui représentent donc un nouveau niveau d’organisation structural de la chromatine intermédiaire entre les nucléosomes et les TADs. Des études futures devraient permettre de mieux comprendre les mécanismes moléculaires impliqués dans leur formation ainsi que leurs rôles fonctionnels dans la régulation du génome.
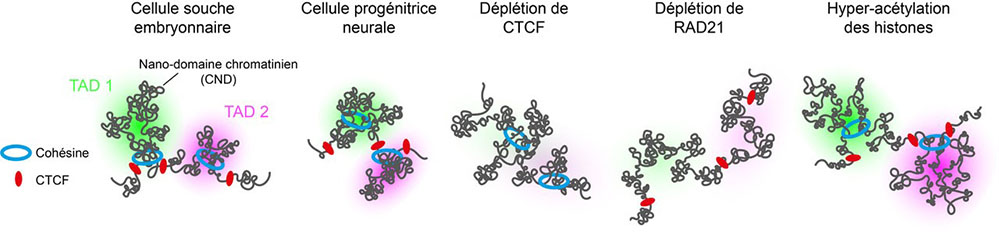
Figure :
Représentation de l’organisation des chromosomes en TADs. Les frontières des TADs sont occupées par la protéine CTCF, tandis que le complexe cohésine génère des interactions chromatiniennes au sein des TADs. Les TADs sont sous-divisés en nano-domaines chromatiniens (CNDs) qui se forment par agrégation de nucléosomes. La ségrégation spatiale des TADs est renforcée lors de la différentiation des cellules souches embryonnaires en cellules progénitrices neurales. Lors de la déplétion de CTCF, le complexe cohésine peut continuer à agir à travers les frontières entre les TADs, induisant alors des contacts ectopiques entre TAD voisins. Lors de la déplétion de RAD21, les interactions chromatiniennes générées au sein des TADs sont abolies. Suite à l’hyper-acétylation des histones, les TADs restent spatialement séparés mais leur organisation interne en CNDs est perturbée.
Pour en savoir plus :
Regulation of single-cell genome organization into TADs and chromatin nanodomains.
Szabo Q, Donjon A, Jerković I, Papadopoulos GL, Cheutin T, Bonev B, Nora EP, Bruneau BG, Bantignies F, Cavalli G.
Nat Genet. 2020 Oct 19. doi: 10.1038/s41588-020-00716-8.
Contact
Laboratoire
Institut de Génétique Humaine (IGH) - (CNRS/Université de Montpellier)
141, rue de la Cardonille
34396 Montpellier - Cedex 5, France