Vers une meilleure compréhension des mécanismes moléculaires qui contrôlent la fertilité.
À l’intérieur de chaque noyau cellulaire, l’ADN est compacté autour d’un assemblage de protéines sous forme de chromatine. L’établissement de la structure correcte de la chromatine au cours de l’oogenèse est indispensable à la fertilité. La protéine LSD1 est un régulateur clé de la chromatine et son absence est responsable d’infertilité. Cependant, les fonctions de LSD1 dans les ovaires sont mal caractérisées. Des chercheurs travaillant sur la drosophile ont découvert qu’au cours de l’oogenèse, LSD1 régule des gènes cruciaux pour le développement et réprime les éléments transposables, ces séquences d’ADN mobiles dont l’activation peut entrainer la stérilité. Cette étude est publiée dans la revue Nucleic Acids Research.
Chaque cellule d'un organisme contient la même information génétique, mais seul un sous-ensemble de gènes est transcrit dans une cellule donnée à un moment donné. La structure de la chromatine est au cœur de la régulation de cette transcription spécifique à la cellule. L'unité de base de la chromatine est le nucléosome, qui est constitué d'ADN enroulé autour d’un assemblage d’histones. Les histones font l'objet d'un grand nombre de modifications post-traductionnelles (PTM) dont la régulation dynamique par des enzymes spécifiques est critique pour la plasticité de la chromatine. Ainsi, l'histone déméthylase LSD1, qui est apparue ces dernières années comme un régulateur clé de la chromatine, joue un rôle essentiel dans la régulation de la différenciation cellulaire et est surexprimée dans certains cancer. Plusieurs inhibiteurs de LSD1 ont été développés et ont été utilisés avec succès contre ces formes de cancer.
LSD1 est responsable de la déméthylation des histones et joue un rôle important dans le contrôle de l'expression des gènes en tant que co-activateur ou co-répresseur transcriptionnel. La protéine LSD1 est évolutivement très conservée et les chercheurs ont montré que l'élimination de son homologue (dLsd1) chez la mouche du vinaigre, Drosophila melanogaster, affecte fortement la fertilité. Cependant, les fonctions spécifiques de dLsd1 dans l'oogenèse sont actuellement mal caractérisées.
Afin de mieux définir son rôle dans les ovaires, les chercheurs ont réalisé des études par immunoprécipitation de chromatine suivie de séquençage à l'échelle du génome (ChIP-seq) permettant de déterminer la localisation de dLsd1 sur l’ADN. Ces analyses, couplées à une étude des changements dans l’expression des gènes (transcriptome) lors de la déplétion de dLsd1, montrent que dLsd1 contrôle certains gènes clés du développement. Ils ont en outre découvert une interaction inattendue entre dLsd1 et le facteur de transcription GATA Serpent, un régulateur important de l'hématopoïèse, pour lequel est mis en évidence pour la première fois un rôle dans l’oogenèse.
Les données transcriptomiques montrent par ailleurs que la réduction des niveaux de dLsd1 entraîne l'expression ectopiques d'éléments transposables (TE). Les TEs sont des éléments génétiques mobiles qui occupent une grande partie des génomes eucaryotes. En raison de leur mobilité, ils constituent des menaces potentielles pour le génome de l'hôte et leur activation a été associée à l'infertilité. Il est donc important de comprendre comment les TEs sont régulés au niveau de la chromatine. Dans cette étude, les chercheurs montrent que la réduction de l’expression de dLsd1 modifie la structure de la chromatine au niveau des éléments transposables et que dLsd1 interagit avec la protéine argonaute, Piwi, un régulateur clé de la répression des TEs.
En résumé, ce travail fournit des informations importantes sur les mécanismes moléculaires par lesquels l'histone déméthylase dLsd1 contrôle l’oogenèse chez la drosophile. Cette étude ouvre la voie à une meilleure compréhension de ces mécanismes et de leur rôle potentiel dans la fertilité chez les mammifères.
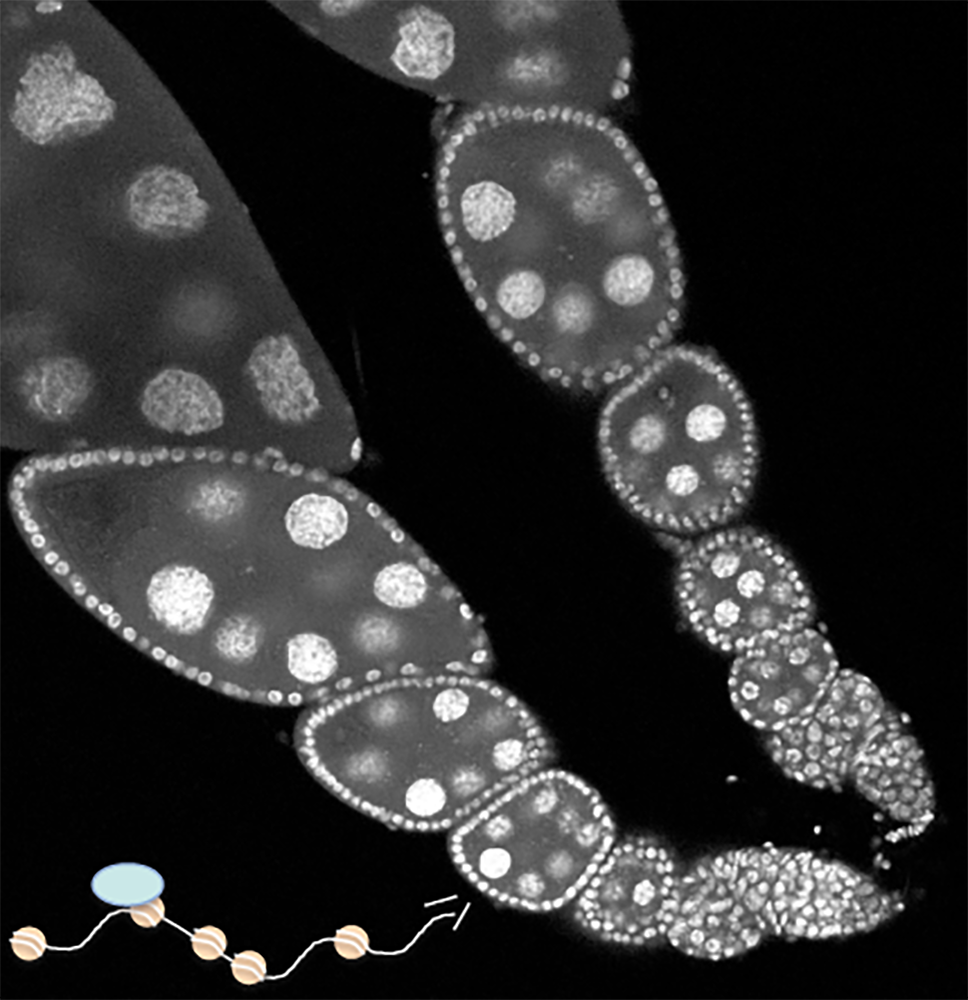
Figure : Image des deux ovarioles, composants des ovaires de la drosophile. Chaque ovariole comprend une succession de groupes de cellules à différents stades de développement (cellules visualisées ici par coloration de leur noyau en blanc). Sur le schéma en bas à gauche, l’ADN (trait blanc) des cellules de l’ovariole est compacté et son expression régulée par les histones (en rose) et par des protéines comme dLsd1 (en bleu).
Pour en savoir plus :
A dual role of dLsd1 in oogenesis: regulating developmental genes and repressing transposons.
Lepesant JMJ, Iampietro C, Galeota E, Augé B, Aguirrenbengoa M, Mercé C, Chaubet C, Rocher V, Haenlin M, Waltzer L, Pelizzola M, Di Stefano L.
Nucleic Acids Res. 2019 Dec 4. pii: gkz1142. doi: 10.1093/nar/gkz1142. [Epub ahead of print]