Christian LesterlinMicrobiologie Moléculaire et Biochimie Structurale (MMSB) - CNRS / Université de Lyon
Mes recherches
J’ai obtenu un doctorat en génétique bactériennes et microbiologie de l’Université de Toulouse (Cornet LAB, LMGL) en 2005. J’ai ensuite réalisé deux expériences postdoctorales (Boccard LAB, I2BC-2006/2009 ; Sherratt LAB, Université d’ Oxford-2009/2015). Mes recherches portaient sur l’étude des mécanismes fondamentaux du cycle cellulaire bactérien, en particulier sur la coordination entre réplication et la ségrégation du chromosome et la division cellulaire. J’ai développé une expertise en analyse multi-échelles des populations bactériennes, combinant la microscopie à haute résolution en cellules vivante (live 4D-SIM), la cytométrie et des essais génétiques. L’obtention du financement ATIP-Avenir m’a permis de créer mon équipe de recherche ou nous appliquons ces approches à l’étude du transfert d’ADN entre cellules bactériennes. Notre objectif est de mieux comprendre la propagation des résistances aux antibiotiques et de la pathogénie dans le monde bactérien.
Mon projet ATIP-Avenir
Acquisition of pathogenesis by horizontal gene transfer in bacteria and intracellular organization of bacterial pathogens
Comprendre comment une bactérie commensale devient pathogène ou résistante aux antibactériens est un enjeu majeur de la microbiologie moderne. Au laboratoire, nous étudions l’acquisition d’ADN exogène par transfert horizontal de gènes (HGT) à l’échelle de la cellule et de la population. Nous nous intéressons particulièrement à la conjugaison bactérienne, principal mécanisme impliqué dans le transfert d’ADN entre cellules en contact direct. Notre stratégie expérimentale combine génétique moléculaire et microscopie en cellules vivantes et permet la visualisation des étapes clés de la conjugaison intra- et inter-espèces. Nous caractérisons la chronologie et la dynamique du transfert de l’ADN, ainsi que l’établissement des propriétés métaboliques associées, comme la pathogénicité ou la multi-résistance aux antibiotiques.
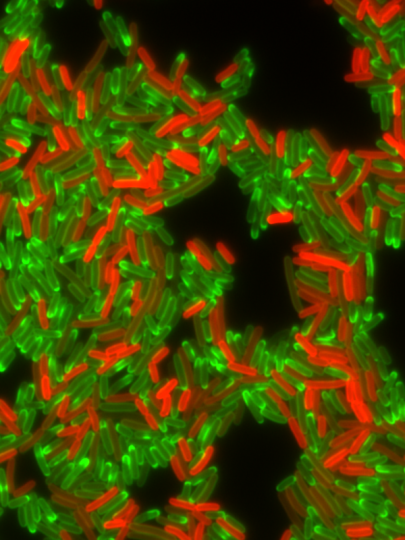
Légende de l'image
Population of drug resistant bacterial cells producing a fluorescent labelled resistance factor, TetA efflux pump (in red), during treatment with tetracycline antibiotic (in green). Cell producing high levels of TetA efflux pump a capable or effective drug efflux and contain very little tetracycline drug. By contrast, cells containing a lot of tetracycline fail to produce TetA efflux pump.