Voyage moléculaire au cœur de l’expression génétique mitochondriale
La mitochondrie, organite cellulaire, possède son propre génome qui produit les ARN essentiels à la production de protéines de la chaine respiratoire qui fournit l’énergie à nos cellules. Les altérations dans cette production sont à l’origine de pathologies graves. Dans un article publié dans Nature communications, des scientifiques visualisent à l’échelle moléculaire les étapes de maturation des ARN mitochondriaux.
Le génome mitochondrial humain transcrit ses propres ARN.
Les mitochondries jouent un rôle essentiel dans nos cellules en convertissant le glucose, issu de la digestion des nutriments, en ATP, une forme d’énergie directement utilisable par la cellule. Ce processus se déroule dans la chaîne respiratoire située dans les mitochondries. Les mitochondries sont des organites cellulaires qui contiennent leur propre génome. Ce dernier doit être transcrit et traduit pour produire des protéines mitochondriales essentielles à cette production d’énergie. Le génome mitochondrial code uniquement pour les ARN nécessaires à la traduction de ces protéines essentielles de la chaine respiratoire : les ARN messagers (ARNm), ARN de transfert (ARNt) et ARN ribosomiques (ARNr). Toutes les protéines nécessaires à la traduction sont encodées dans le génome du noyau de la cellule, transcrites dans le noyau, traduites dans le cytoplasme et importées dans la mitochondrie.
Le génome mitochondrial humain est ainsi transcrit en deux longues molécules d’ARN, une pour chaque brin, contenant des ARNm, des ARNr et des ARNt, tous destinés à produire des protéines essentielles de la chaîne respiratoire. L'excision précise des ARNt par des enzymes, les ribonucléases mitochondriales, mt-RNase P et Z, libère tous les différents types d'ARN contenus dans ces deux molécules d’ARN. Les ARNt subissent ensuite l’addition de la séquence CCA à leur extrêmité 3’, processus crucial pour leur bon fonctionnement dans la traduction mitochondriale.
Décrypter les mécanismes pour comprendre les maladies mitochondriales.
Les altérations de la maturation des mt-ARNt sont néfastes sur l'expression des gènes et l'assemblage des ribosomes dans l'organite et les mutations qui perturbent les étapes de maturation des mt-ARNt ou leurs structures sont responsables de maladies humaines graves.
Dans un article publié dans la revue Nature Communications, les scientifiques ont utilisé la cryo-microscopie électronique(CryoEM), pour visualiser à l’échelle atomique les structures des machineries moléculaires responsables de quatre étapes clés de maturation des ARNt mitochondriaux humains.
Ces travaux montrent comment une enzyme de modification, en plus d’ajouter un méthyle sur les mt-ARNt, accompagne les mt-ARNt, à la structure tri-dimensionnelle « fragile », tout au long de leur maturation. Ils expliquent aussi pourquoi ces étapes de maturation se déroulent dans un certain ordre. En effet, ils montrent que les mt-ARNt sont d’abord pris en charge pour un complexe méthyltransférase d’ARNt, constitué des protéines TRMT10C/SDR5C1. C’est sur ce complexe que les enzymes de maturation vont venir chacune leur tour modifier les extrémités des mt-ARNt pour les rendre fonctionnels pour la traduction mitochondriale. La première enzyme à maturer les mt-ARNt est la RNase P, dont l’activité est portée par la protéine PRORP, qui n’est active que lorsque les mt-ARNt sont fixés au complexe méthyltransférase. Une interaction entre PRORP et TRMT10C permet à la protéine PRORP de positionner son site actif sur l’extremité des mt-ARNt à traiter. Sans cette interaction, PRORP n’est pas ou très peu active. PRORP laisse ensuite la place à ELAC2 pour traiter la deuxième extrêmité des mt-ARNt, l’extrêmité 3’. ELAC2 ne peut intervenir que lorsque PRORP a traité l’extrêmité 5’, du fait du manque de place pour que les deux enzymes interagissent en même temps sur les mt-ARNt et parce qu’ELAC2, contrairement à PRORP, n’est pas active sur un mt-ARNt avec une extrêmité 5’ non traitée par PRORP. ELAC2 laisse ensuite la place à TRNT1 qui va ajouter la séquence CCA en 3’ des mt-ARNt, ce qui prépare l’extrêmité 3’ à l’ajout d’un acide aminé par les aminoacyl-ARNt synthétases.
Ce travail fournit ainsi une « cinématographie moléculaire » du processus de maturation des ARNt mitochondriaux. La visualisation à l’échelle atomique de ces complexes enzymatiques fonctionnels a répondu à de nombreuses questions non résolues depuis des décennies et ouvrent de nouvelles perspectives, notamment pour des applications thérapeutiques dans le cadre du traitement du cancer.
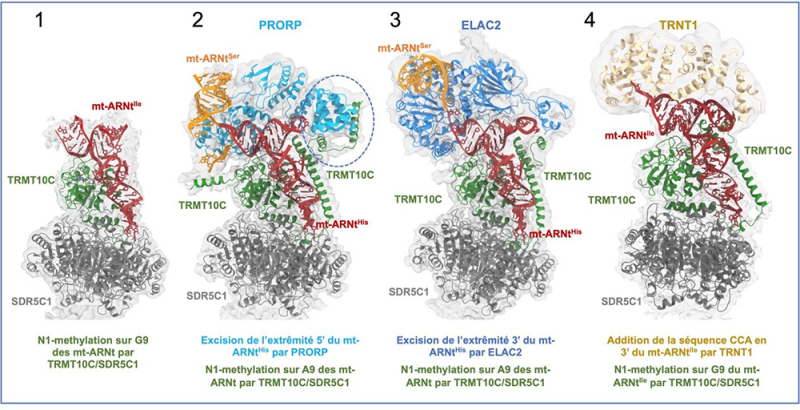
Figure : Structures cryoEM des complexes de maturation des ARNt mitochondriaux humains montrant la chronologie des évènements de maturation : 1) la N1-méthylation de G9 du mt-ARNtIle par le complexe méthyltransférase TRMT10C/SDR5C1, 2) l’excision de l’extrêmité 5’ du mt-ARNtHis par PRORP et la N1-méthylation de A9 du mt-ARNtHis par le complexe méthyltransférase TRMT10C/SDR5C1, 3) l’excision de l’extrêmité 3’ du mt-ARNtHis par ELAC2 et la N1-méthylation de A9 du mt-ARNtHis par le complexe méthyltransférase TRMT10C/SDR5C1, 4) l'ajout de la séquence CCA en 3’ du mt-ARNtIle par TRNT1 et la N1-méthylation de G9 du mt-ARNtIle par le complexe méthyltransférase TRMT10C/SDR5C1. Les densités électroniques sont représentées par les surfaces en gris clair et les modèles ajustés dans les densités sont indiqués. Le cercle en pointillé indique l'interaction entre PRORP et TRMT10C nécessaire pour le clivage de l'extrémité 5' du mt-ARNt par PRORP.
En savoir plus : Meynier, V., Hardwick, S.W., Catala, M. et al. Structural basis for human mitochondrial tRNA maturation. Nat Commun 15, 4683 (2024). https://doi.org/10.1038/s41467-024-49132-0
Contact
Laboratoire
Institut de biologie physico-chimique – IBPC (CNRS)
Laboratoire d’expression génétique microbienne
13 rue Pierre et Marie Curie,
75005 Paris