Visualisation à haute résolution des modifications chimiques et des ions dans la structure du ribosome humain
Dans un article publié dans la revue Nature Structural Molecular Biology, des scientifiques décrivent les modifications chimiques et le rôle des ions et molécules d’eau au sein du ribosome humain mature. L’environnement structurale de ces modifications ARN apporte des informations inédites pour comprendre l’architecture du ribosome humain, visualisé à mieux de 2 Å de résolution par cryo microscopie électronique. Ceci ouvre la porte à la compréhension de certaines pathologies tel que le développement tumoral qui dépend d’une forte synthèse protéique.
La biogenèse des ribosomes est un processus fondamental complexe permettant la formation des ribosomes, organites qui assurent la production des protéines. On sait en particulier que cette biogenèse est suractivée dans les cellules cancéreuses afin d’assurer l’augmentation de leur prolifération.
De nombreuses nouvelles modification de l’ARN ribosomal mises en évidence
Dans un article publié dans la revue Nature Structural and Molecular Biology, les scientifiques décrivent les modifications chimiques qui se produisent sur l’ARN du ribosome au cours de sa production. Grâce aux technologies de cryo-microscopie électronique de dernière génération et en combinaison avec un traitement d'images avancé pour trier les différents états structuraux, et en poussant la résolution par des classifications et des affinements concentrés sur différentes régions de l'objet biologique d'intérêt, il est devenu possible de franchir la barrière de résolution de 2 Å pour le ribosome humain 80S entièrement assemblé, atteignant 1.7 Å de résolution localement. Ce travail a permis de montrer de nouvelles modifications de l’ARN ribosomique et des ions fonctionnellement importants tels que les ions Zinc (Zn2+), Potassium (K+) et Magnésium (Mg2+). Grace aux techniques de pointe utilisées, les chercheurs ont pu observer ces ions avec leurs molécules d’eau associées. En utilisant en parallèle une technologie de spectrométrie de masse sur des fragments d’ARN ribosomique obtenus après digestion enzymatique leur localisation au sein du ribosome a pu être confirmé.
Les mécanismes moléculaires sous-jacents dévoilés
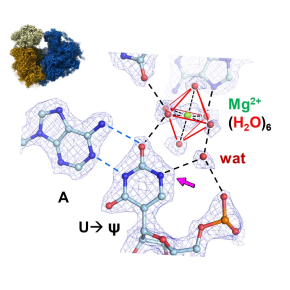
L'approche intégrée de l'analyse structurale à haute résolution, de l'analyse approfondie par spectrométrie de masse et du séquençage de l'ARNr a favorisé une synergie unique entre l'analyse structurale et biochimique des modifications chimiques de l'ARNr, permettant une analyse complète de leur environnement tridimensionnel au niveau moléculaire. La méthylation du groupement ribose 2'-OH de l'ARNr crée non seulement une face hydrophobe qui facilite les contacts de van der Waals, mais modifie également le schéma de liaisons hydrogènes en faveur d'un rôle d'accepteur uniquement. L'isomérisation des uridines en pseudo-uridines (ψ) introduit une position polaire à la position N1 de la base nucléotidique qui génère de nouvelles possibilités de liaison hydrogène avec les résidus voisins, par exemple avec une molécule d'eau pontante, avec un ribose ou avec un groupement phosphate de l'ARN. La visualisation des ions (Zn2+, K+ et Mg2+) et des molécules d'eau qui leur sont associées (par exemple sous la forme d’octaèdres de Mg2+ hexa-hydratées, avec une substitution variable par les acides aminés et acides nucléiques du ribosome) est un autre élément clé de ce travail, qui révèle que les ions Mg2+ n'interagissent pas nécessairement directement avec les groupes de phosphate de l'ARN, mais que de nombreuses interactions sont médiées par les molécules d'eau, maintenant de manière flexible la structure ARN-protéine de la machinerie de traduction humaine.
L'ensemble des résultats obtenus donne un aperçu sans précédent de la localisation, de l'environnement 3D et du rôle des modifications chimiques dans le ribosome humain 80S entièrement assemblé, dans les sous-unités ribosomiques 40S et 60S individuelles et dans leurs régions d'interface. Un aspect particulièrement intéressant est le rôle des pseudo-uridines : dans le cas d'une conversion de la paire de bases A-U en A-ψ, la liaison H supplémentaire compense les interactions plus faibles de la paire de bases par rapport à une paire G-C plus stable (3 liaisons hydrogène). Ce principe de stabilisation des paires de bases A-Ψ établit un concept important applicable à l'ARN en général. La structure met également en évidence le rôle des ions et des interactions médiées par les molécules d'eau dans la stabilisation de l'architecture de l'ARN, avec des implications pour le repliement de l'ARN et du point de vue de la chimie de coordination et de la catalyse dans les assemblages macromoléculaires.
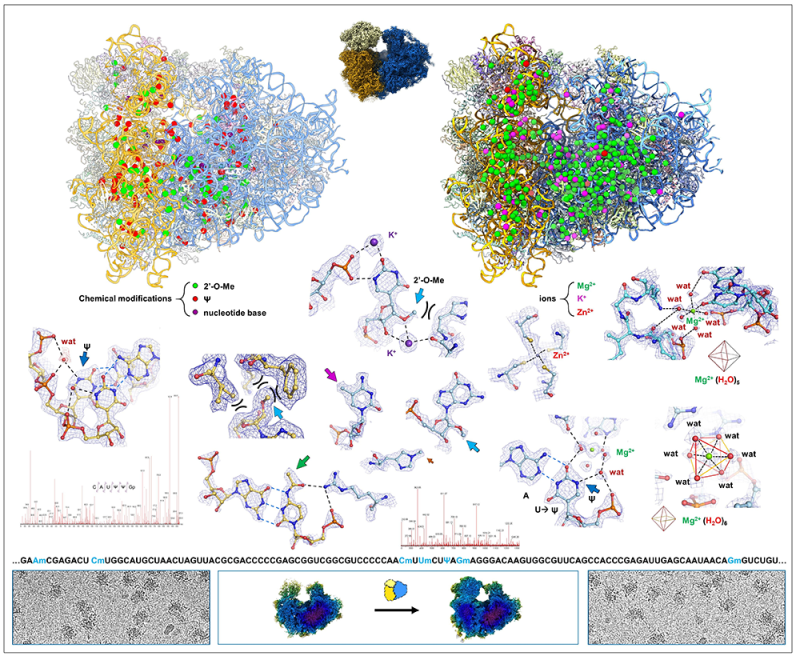
Figure : Détails de la structure cryo-EM du ribosome humain à une résolution de 1.9 Å. Structure du ribosome humain (en haut au milieu) avec indication des positions des modifications chimiques (en haut à gauche) ou des ions (en haut à droite), résolu à une résolution de 1.9 Å de résolution et atteignant 1.7 Å localement. Les vues détaillées ci-dessous montrent des exemples de différentes régions et résidus (nucléotides et acides aminés) comportant des modifications visibles sur la carte cryo-EM (partie gauche) et validées par spectrométrie de masse (2 spectres sont montrés pour illustration) ; les modifications post-traductionnelles des protéines ribosomiques sont également visibles (milieu) ; les ions et leur environnement, y compris les molécules d'eau associées aux ions Mg2+ (en coordination octaédrique) et à la position N1 des pseudo-uridines, fournissant des liaisons hydrogène supplémentaires pour la stabilisation des conversions de paires de bases A-U en A-ψ dans l'architecture de l'ARN.
En savoir plus : Chemical modifications, ions and water molecules in the sub-2 Å resolution structure of the human 80S ribosome. Nat Struct Mol Biol (2024).
https://doi.org/10.1038/s41594-024-01275-w.
Contact
Laboratoire
Institut de génétique et de biologie moléculaire et cellulaire - IGBMC (CNRS/Inserm/Université de Strasbourg)
1, rue Laurent Fries
67404 ILLKIRCH CEDEX