Un nouvel acteur dans le bourgeonnement du virus du SIDA
Pour en savoir plus
Dans une étude publiée dans la revue Nature Communications, des scientifiques révèlent l’existence de nouveaux antigènes produits à partir du génome du virus et capables de générer de puissantes réponses immunitaires. Cette découverte élargit les cibles potentielles des vaccins contre le VIH et pourrait ouvrir la voie à de nouvelles stratégies thérapeutiques.
Les lymphocytes T jouent un rôle crucial dans le contrôle de l’infection par le virus de l’immunodéficience humaine-1 (VIH). Leur activation et la mise en place d’une réponse immunitaire dépendent de la reconnaissance de peptides viraux, appelés aussi antigènes, présentés à la surface des cellules infectées par l’intermédiaire des molécules du complexe majeur d’histocompatibilité (CMH). Ces antigènes proviennent de protéines virales produites lors du cycle d’infection de la cellule hôte par le VIH.
Les protéines virales sont générées par la machinerie de traduction cellulaire qui transforme les ARN messagers (ARNm) en protéines. Les ribosomes cellulaires traduisent les ARNm du VIH à partir de cadres de lecture ouverts (ORF), qui sont des segments d’ARNm contenant l’information nécessaire pour produire une protéine. Il est couramment admis que les antigènes du VIH proviennent uniquement de protéines dites classiques du VIH.
Cependant, des progrès récents en génomique et en peptidomique ont remis en question cette définition des ORF. Des milliers de petites ORF (small ORF ou sORF) codent pour des polypeptides ou des microprotéines de moins de 100 acides aminés. Ces sORF sont largement répartis le long du génome humain, certains chevauchent les ORF classiques sur un cadre différent appelé cadre de lecture alternatif (ARF), mais la majorité se trouve dans les régions en amont (UTR) des gènes connus.
Il a été estimé que 85 % des produits de traduction proviennent de régions non annotées du génome humain et principalement de séquences hors cadre de l'ORF. Des études récentes ont révélé qu'une fraction des peptides présentés par les molécules du CMH est dérivée de polypeptides codés par des sORF qui sont spécifiques ou surreprésentés dans les cellules tumorales.
Dans le contexte du VIH-1, des recherches antérieures ont montré que les lymphocytes T spécifiques du VIH ciblent également les peptides dérivés des ARF du VIH présentés par les molécules du CMH. Cependant, l'existence d'ARF dans le génome du VIH n’a été démontrée jusqu’à présent que par des approches indirectes.
Dans une étude publiée dans la revue Nature Communications, les scientifiques ont utilisé une technique appelée profilage des ribosomes (Riboseq) pour identifier toutes les séquences d’ARNm du VIH-1 qui sont activement traduites en protéines dans les cellules T CD4+ infectées. En bloquant la traduction qui se déroule au sein des ribosomes et en séquençant les ARN restés accrochés, cette approche leur a permis de cartographier systématiquement ce qu’on appelle le « translatome » du VIH-1, c’est-à-dire l’ensemble des ARN viraux en cours de traduction dans les cellules infectées. Ils ont ainsi découvert que le génome du VIH-1 contient une centaine d’ARF correspondant à des sORF, situées soit en amont des gènes codant les protéines virales classiques, soit chevauchant les régions codantes principales du VIH.
De manière remarquable, en utilisant deux approches indépendantes et complémentaires, les scientifiques ont démontré que les ARF du VIH codent pour de nouveaux antigènes viraux capables d'induire des réponses larges et puissantes des lymphocytes T. Ces résultats élargissent le spectre des antigènes du VIH qui pourraient être utilisés à la conception de vaccins. Ils pourraient également mettre en évidence l’existence de microprotéines ou de pseudogènes du VIH.
Dans une autre publication parue dans le même numéro de Nature Communications, les scientifiques ont étudié les fonctions biologiques des ARF situés dans les régions 5’UTR des gènes codant pour les protéines virales. Ils démontrent que ces ARF exercent une action majeure pour le virus en contrôlant la traduction des protéines virales classiques. Ces ARF codent également pour des antigènes viraux reconnus par le système immunitaire.
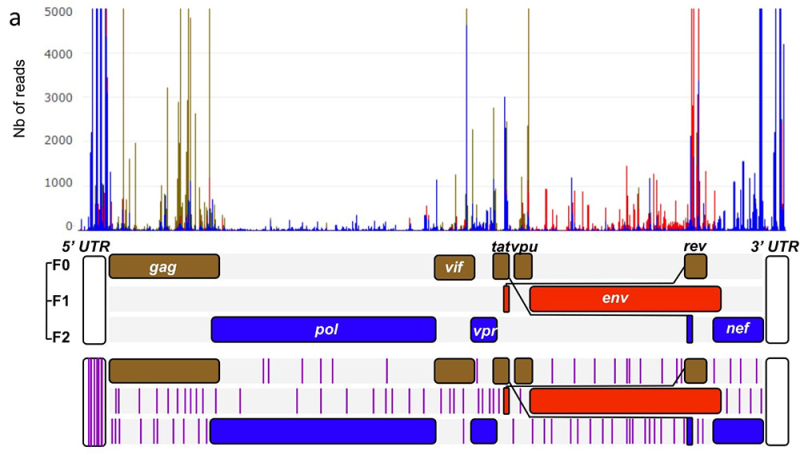
Figure : Le paysage translationnel du VIH-1 : identification des ARFs. (a) Distribution des fragments protégés par des ribosomes (RPF) dans le génome du VIH-1 obtenus à partir de cellules infectées par HIV-1. Le nombre de RPF (Nb of reads) est représenté en fonction de la position dans le génome du HIV-1 (NL4-3). Les RPF traduits dans les phases de lecture 0, +1 et +2 sont représentés respectivement en marron, rouge et bleu. L'ARN génomique du VIH est représenté ci-dessous ; les cadres de lecture canoniques du VIH-1 sont indiqués et colorés en fonction de leurs phases de lecture. Le positionnement des ARF (lignes horizontales violettes) est indiqué dans la représentation inférieure du génome de l’ARN. Ces ARF, fortement conservés parmi les clades du VIH-1, codent pour des peptides immunogéniques reconnus par des cellules T isolées de personnes vivant avec le VIH-1.
Référence : Unveiling conserved HIV-1 open reading frames encoding T cell antigens using ribosome profiling. Bertrand L, Nelde A, Ramirez BC, Hatin I, Arbes H, François P, Demais S, Labaronne E, Decimo D, Guiguettaz L, Grégoire S, Bet A, Beauclair G, Gross A, Ziegler MC, Pereira M, Jeger-Madiot R, Verdier Y, Vinh Y, Cardinaud S, Graff-Dubois S, Esclatine A, Gouttefangeas C, Altfeld M, Hocqueloux L, Samri A, Autran B, Lambotte O, Rammensee H-G, Ricci EP, Walz W, Namy O and Moris A.
Nature Communications, 18 février 2025, DOI : https://doi.org/10.1038/s41467-025-56773-2
Voir aussi : Extensive uORF translation from HIV-1 transcripts conditions DDX3 dependency for expression of main ORFs and elicits specific T cell immune responses in infected individuals. Labaronne E, Décimo D, Bertrand L, Guiguettaz L, Sohier TMJ, Cluet D, Vivet-Boubou V, Dahoui C, François P, Hatin I, Lambotte O, Samri A, Autran B, Etienne L, Goujon C, Paillart J-C, Namy O, Ramirez BC, Ohlmann O, Moris A and Ricci EP. 2025.
Nature Communications, 18 Fevrier 2025, DOI : https://doi.org/10.1038/s41467-025-56772-3
Pour en savoir plus
Institut de biologie intégrative de la cellule - I2BC (CNRS/CEA/Université Paris-Saclay)
Équipe Autophagie et Immunité Antivirale
1 Avenue de la Terrasse
91198 Gif-sur-Yvette - FRANCE