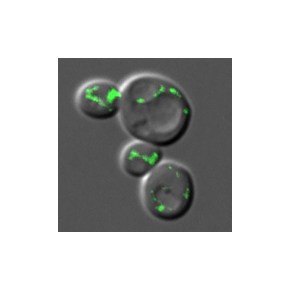
Une GFP fragmentée bi-génomique pour étudier les protéines multi-localisées
Les scientifiques ont créé une souche de levure exprimant une GFP (Green Fluorescent Protein) fragmentée bi-génomique (codée par les génomes nucléaire et mitochondrial) permettant de rendre fluorescente uniquement la fraction mitochondriale de protéines se localisant simultanément dans la mitochondrie et dans un autre compartiment cellulaire. Ces résultats sont publiés dans la revue Elife.
De nombreuses études requièrent l'étude de la localisation de protéines d'intérêt au sein d'une cellule. En fusionnant la protéine étudiée à la GFP (Green Fluorescent Protein), une protéine fluorescente de méduse, elle devient fluorescente et il est alors possible d’observer sa localisation subcellulaire par microscopie. Cependant, cette méthode présente, chez les eucaryotes, des difficultés liées à leur structure même. Les eucaryotes sont en effet des organismes dont les cellules possèdent un noyau (le "génome nucléaire" renfermant la majorité des gènes de l'organisme) mais aussi des mitochondries possédant leur propre ADN (le "génome mitochondrial)", indépendant du génome nucléaire et qui ne renferme qu’un très petit nombre de gènes.
Les mitochondries, au sein desquelles sont réalisés de nombreux processus cellulaires essentiels, dont la respiration, sont d’anciennes bactéries symbiotiques dont la quasi-totalité des gènes a été transférée dans le génome nucléaire au cours de l'évolution. Cela signifie que la très grande majorité des protéines mitochondriales est codée par des gènes nucléaires, donc synthétisées dans le cytoplasme, avant d’être transportées dans la mitochondrie. Cela est possible parce que ces protéines mitochondriales codées dans le noyau possèdent une courte séquence protéique d’adressage reconnue par une machinerie moléculaire de transport qui permet de les reconnaître et de les faire passer à l’intérieur de l’organite.
Cependant, de nombreuses études ont montré que 30 % des protéines mitochondriales sont, en réalité, localisées à la fois dans la mitochondrie et un autre compartiment (cytoplasme, noyau…) On parle de « double adressage » subcellulaire et de protéines doublement localisées. Il est donc extrêmement difficile de visualiser la fraction mitochondriale d’une protéine fluorescente doublement localisée de ce type, parce que la fluorescence de la fraction cytoplasmique éclipse généralement celle de la fraction mitochondriale.
Pour résoudre ce problème, les scientifiques ont développé une souche de levure exprimant une GFP fragmentée, ou Split-GFP, dont les deux fragments sont exprimés à partir de deux génomes distincts, nucléaire et mitochondrial (on peut dire qu'elle est "bi-génomique"). Ainsi, pour étudier une protéine doublement localisée, on modifie son gène de sorte qu’une fois synthétisée dans le cytoplasme, elle est fusionnée à un fragment seulement de la GFP (GFPß11), et n’est pas fluorescente. L’idée est de la rendre fluorescente uniquement lorsqu’elle rejoint l’intérieur de la mitochondrie. Pour cela, il faut que le second fragment de la GFP (GFPß1-10) se trouve déjà à l’intérieur de la mitochondrie et jamais en dehors. L’innovation consiste à modifier l’ADN mitochondrial de sorte que ce second fragment de la GFP soit exprimé à partir du génome mitochondrial lui-même. Ainsi, lorsqu’une protéine doublement localisée est ainsi marquée, elle ne devient fluorescente que dans la mitochondrie, grâce à la reconstitution d’une GFP complète. On s'affranchit ainsi de la fluorescence "parasite" de la fraction non mitochondriale de la protéine doublement localisée.
Cette souche de levure pourrait maintenant servir de plateforme de criblage de « l’importabilité » mitochondriale de protéines de levure mais également de plantes ou humaines. Cet outil leur a déjà permis de mettre en évidence l’import mitochondrial de protéines qui étaient jusqu’alors considérées comme uniquement cytoplasmiques et devrait permettre des avancées marquantes dans l’analyse de mitoprotéomes.
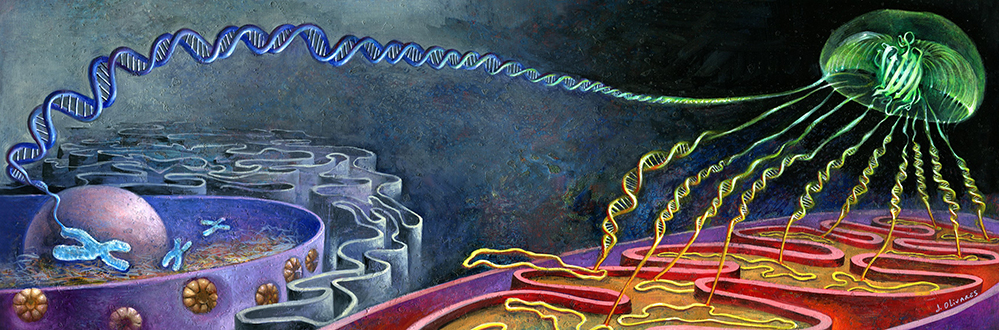
Figure : Une peinture métaphorique de l’artiste Jaime Olivares illustrant l’origine Bi-génomique de la GFP fragmentée. Le fragment GFPß1-10 non fluorescent comprenant les 10 premiers brins ß de la GFP de la méduse Aequorea victoria est exprimé uniquement par l’ADN mitochondrial alors que le fragment GFPß11 lui aussi non fluorescent fusionné à la protéine dont on veut visualiser la localisation mitochondriale est exprimée par l’ADN nucléaire. Si la protéine fusionnée au fragment GFPß11 se localise dans la mitochondrie les 2 fragments s’auto-assemblent pour reconstituer une GFP active qui rend uniquement les mitochondries fluorescentes.
Pour en savoir plus:
Assigning mitochondrial localization of dual localized proteins using a yeast Bi-Genomic Mitochondrial-Split-GFP
Bader, G., Enkler, L., Araiso, Y., Hemmerle, M., Binko, K., Baranowska, E., De Craene, O. J., Ruer-Laventie, J., Pieters, J., Tribouillard-Tanvier, D., Senger, B., di Rago, J-P., Friant, S., Kucharczyk, R. & Becker, H. D. (2020). Elife 2020; 9:e56649. DOI: https://doi.org/10.7554/eLife.56649.
Contact
Laboratoire
Génétique Moléculaire Génomique Microbiologie (GMGM) - (Université de Strasbourg/CNRS)
Institut de Physiologie et de Chimie Biologique
4, allée Konrad Roentgen
67000 Strasbourg