Une enzyme de plante détournée pour lutter contre des virus
Les virus de plante sont responsables de pertes de rendement agricole considérables à travers le monde. Les scientifiques ont mis au point une nouvelle approche biotechnologique qui consiste à relocaliser dans le cytosol une nucléase de plante afin de couper l’ARN génomique de certains virus et ainsi de bloquer leur réplication. Cette étude est publiée dans la revue Nature communications.
Dans le contexte actuel de changement climatique et de diminution de la surface des terres cultivées, la production de ressources alimentaires suffisantes pour une humanité en croissance rapide est un des défis majeurs du 21ème siècle.
Les virus infectant les plantes (phytovirus) font partie des agents pathogènes les plus dévastateurs et occasionnent des pertes de rendement considérables. Le recours aux biotechnologies ouvre de nouvelles perspectives pour la lutte contre les maladies virales.
Plus de 80% des phytovirus ont un génome à ARN et un grand nombre d’entre eux portent une structure dite « ARNt-like » (TLS) à l’extrémité 3 ’de leur ARN génomique. Cette structure est essentielle pour leur réplication. Les virus contenant un TLS infectent des plantes d'importance économique majeure dans le monde telles que le blé, le maïs, le haricot, la tomate, le chou, le poivron, les cucurbitacées, la pomme de terre, l'arachide ou le cacaoyer.
La RNase P est une enzyme à activité endonucléase responsable de l'une des étapes clés de la maturation des ARN de transfert (ARNt). Au-delà des ARNt, ces enzymes participent également à la maturation de l'ARN messager (ARNm) en clivant des structures TLS présentes aux extrémités de certains ARNm des mitochondries de plante.
Sur la base de cette observation, les scientifiques ont développé une stratégie pour induire une résistance des plantes aux virus contenant un TLS. Ils sont partie de l'hypothèse qu'une enzyme à activité RNase P devrait être capable de cliver la structure TLS des phytovirus, ce qui empêcherait la réplication virale et bloquerait l’infection. Cependant, ce processus ne peut se produire spontanément in vivo car les virus de plante sont situés dans le cytosol alors que les RNase P s’accumulent uniquement dans le noyau, les mitochondries et les chloroplastes. Pour contourner ce problème, les chercheurs ont développé une approche où une RNase P est modifiée pour pouvoir s'accumuler dans le cytosol. L’activité anti-virale de ces RNase P cytosoliques baptisées « CytoRP » est évaluée avec le virus de la mosaïque jaune du navet (TYMV) et le virus de la mosaïque du colza (ORMV). Pour cela, après avoir vérifié la localisation cytosolique de différents variants de CytoRP, des expériences de clivage in vitro ont montré que CytoRP est capable de cliver les structures en TLS du TYMV ainsi que de l’ORMV.
Afin d’évaluer alors cette approche in vivo, des plantes exprimant CytoRP ont été construites par des méthodes transgéniques et cisgéniques (c'est à dire par édition de génome au moyen du système CRISPR-Cas9). La résistance de ces plantes à l’infection virale par le TYMV et l’ORMV a été évaluée. Les résultats, prometteurs, suggèrent que la technologie CytoRP pourrait ouvrir des pistes pour l'amélioration de la protection des cultures et pour un approvisionnement plus sûr en ressources alimentaires, deux préoccupations sociétales majeures.
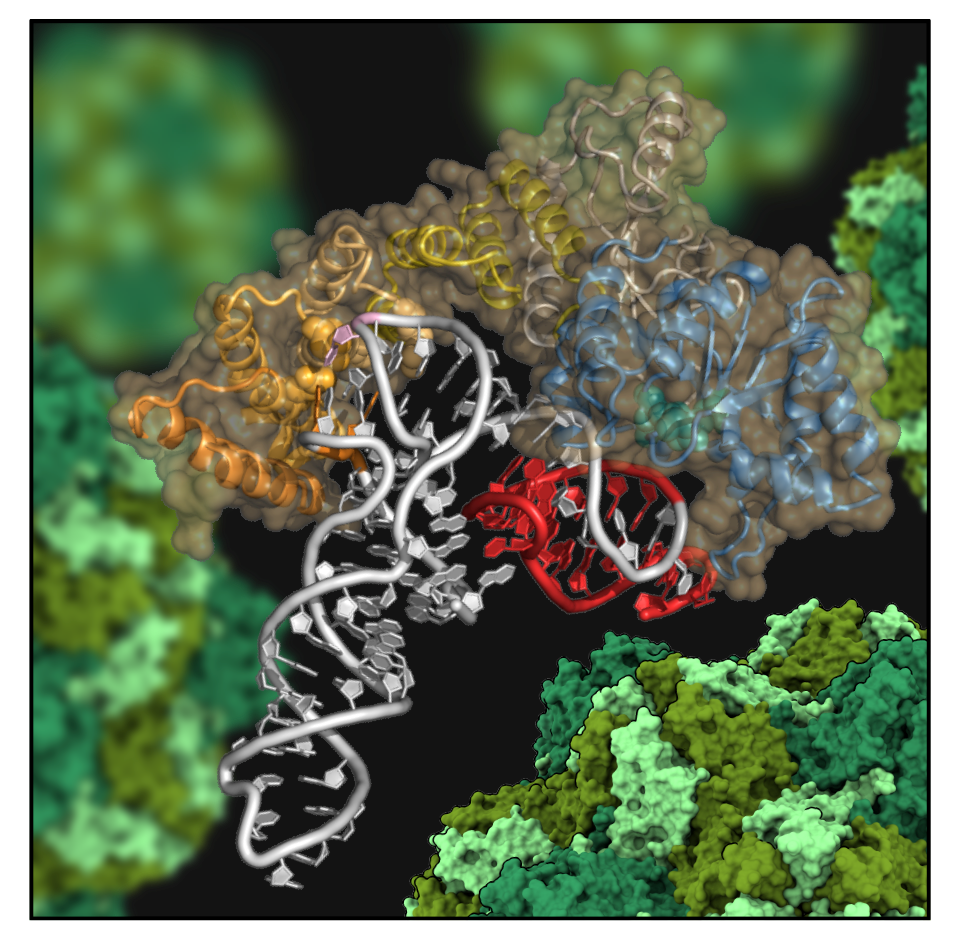
Figure : Modèle structural de la RNase P cytosolique « CytoRP » en interaction avec la structure ARNt-like du virus de la mosaïque jaune du navet. La partie en rouge, représentant l’extrémité 3’ de l’ARN génomique du virus, est coupée par CytoRP, ce qui empêche la réplication du virus d’avoir lieu. Des particules de virus de la mosaïque jaune du navet sont représentées en vert en arrière-plan de la figure.
En savoir plus
Towards plant resistance to viruses using protein-only RNase P.
Gobert A, Quan Y, Arrivé M, Waltz F, Da Silva N, Jomat L, Cohen M, Jupin I, Giegé P.Nat Commun. 2021 Feb 12;12(1):1007. doi: 10.1038/s41467-021-21338-6.
Contact
Laboratoire
Institut de biologie moléculaire des plantes (IBMP) - CNRS
12 Rue du général Zimmer
67084 STRASBOURG CEDEX