Une chorégraphie spatio-temporelle inédite de régulation génétique chez le pneumocoque
Comprendre comment est coordonnée au cours du temps et dans l’espace de la cellule l’expression du potentiel génétique codé dans le génome est une question centrale de la biologie des systèmes. Dans cet article publié dans la revue eLife, cette question a été posée au circuit de régulation qui détermine l’expression transitoire de l’état de compétence chez la bactérie pathogène Streptococcus pneumoniae. Cette étude révèle que les cinq régulateurs maîtres de ce circuit se concentrent à un seul et même pôle de la cellule, définissant ainsi un hub de contrôle inédit du programme génétique de développement de la compétence.
La compétence bactérienne est un état physiologique particulier au cours duquel les cellules développent de nouvelles propriétés, dont la plus célèbre et la plus répandue est la transformation génétique, i.e. un mécanisme de transfert horizontal de gènes impliquant la capture et la recombinaison dans le génome d’ADN exogène.
La compétence se distingue parmi les espèces bactériennes par les mécanismes qui contrôlent son développement. Chez le pathogène majeur de l’homme Streptococcus pneumoniae (le pneumocoque), elle est déclenchée lors de la croissance des cellules en réponse à plusieurs types de stress, dont plusieurs antibiotiques, conduisant à l’induction de l'expression (transcription) d’une centaine de gènes durant une trentaine de minutes. Ce pic d’expression transitoire se décompose en 2 vagues successives et interconnectées. La première vague est activée par un peptide secrété, le CSP ("Competence Stimulating Peptide", codé par le gène comC) qui stimule l’auto-phosphorylation de la kinase membranaire ComD et, en cascade, la phosphorylation du régulateur transcriptionnel ComE. Ce dernier active alors sa propre expression, ainsi que celle de ComD, de ComC et de son système d’export ComAB, créant ainsi une boucle d’auto-activation. ComE phosphorylé (ComE-P) active aussi l’expression d’un facteur sigma alternatif, sX, qui dirige l’ARN polymerase vers les gènes de la seconde vague. Parmi ces gènes se trouve celui qui code pour la protéine DprA, impliquée dans le mécanisme de transformation à l’étape de recombinaison de l'ADN et dans la "fermeture" de la compétence via son interaction spécifique avec ComE-P. Un défaut de DprA dans son rôle régulateur résulte en un allongement de la période de la compétence et une fragilisation de l’intégrité cellulaire.
Pour comprendre comment s’intègre dans la cellule le circuit de régulation de la compétence du pneumocoque, les chercheurs ont étudié par microscopie à fluorescence la dynamique de localisation de DprA, sX, ComE, ComD et du CSP dans les cellules individuelles. De manière remarquable, ComD, le CSP, sX et DprA s’accumulent graduellement au pôle distal des cellules. ComE se concentre au niveau de la membrane en plusieurs amas mobiles localisés temporairement à ce pôle. Étonnamment, l’adressage au pôle de DprA est dépendant de sX, révélant un rôle inédit pour cette sous-unité de l’ARN polymérase en charge de l’initiation de la transcription. Enfin, la fermeture de la compétence est démontrée dépendre de la localisation polaire de DprA. Cette étude montre que le pôle de la cellule constitue un hub de régulation de la compétence du pneumocoque, crucial pour orchestrer son développement transitoire et garantir ainsi le maintien de l’intégrité cellulaire.
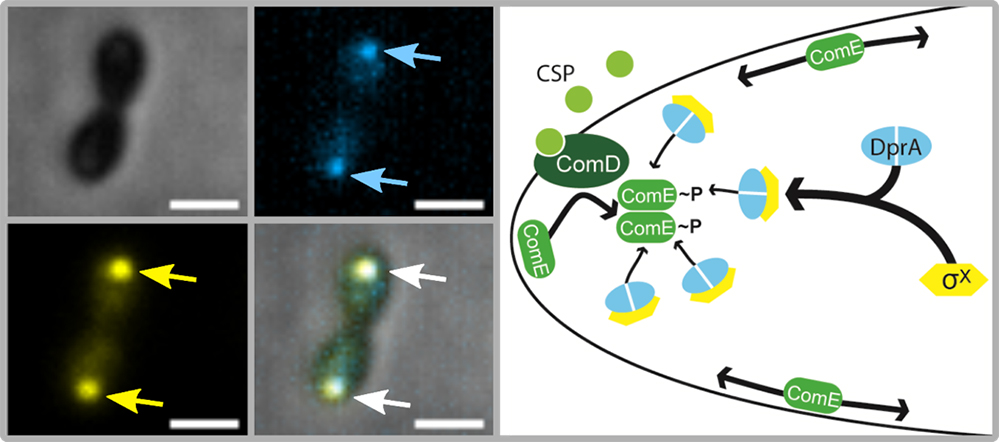
Figure : Le pôle de la cellule définit un hub de régulation de la compétence chez le pneumocoque. (A) Image de cellules montrant la localisation et la co-localisation polaires de σX et de DprA, fusionnées à la YFP (en jaune) et à la mTurquoise (en bleu), respectivement; la barre d’échelle en blanc est de 1mm (B): Modèle de la régulation spatio-temporelle de l'induction et de fermeture de la compétence.
Pour en savoir plus :
The alternative sigma factor σX mediates competence shut-off at the cell pole in Streptococcus pneumoniae.
Johnston CH, Soulet AL, Bergé M, Prudhomme M, De Lemos D, Polard P.
Elife. 2 Nov 2020. DOI: 10.7554/eLife.62907
Contact
Laboratoire
Laboratoire de Microbiologie et de Génétique Moléculaires (LMGM) du Centre de Biologie Intégrative (CBI) de Toulouse - (CNRS/Université Paul Sabatier)
118 route de Narbonne
31062 Toulouse Cedex 9
