Transcription/recombinaison dans la genèse de la diversité des anticorps
L’extraordinaire diversité des anticorps est générée par un processus appelé recombinaison V(D)J qui implique des cassures/fusions de différents segments de gènes distants sur le chromosome. La recombinaison V(D)J est initiée par un complexe protéique appelé RAG. Le modèle qui prévaut actuellement est que la transcription en ARN des différents segments impliqués est absolument requise pour leur recombinaison. Dans cette étude, publiée dans la revue Nucleic Acids Research, les chercheurs montrent que la recombinaison V(D)J peut avoir lieu en l’absence de transcription et que le remodelage de la chromatine des segments suffit au recrutement du complexe RAG pour initier la recombinaison.
Les lymphocytes B jouent un rôle crucial dans la réponse immunitaire adaptative grâce notamment à leur capacité spécifique à produire des anticorps. Les chaînes lourde et légère d’un anticorps sont codées par des loci complexes constitués d’une mosaïque de segments distants les uns des autres. Ces loci subissent une série ordonnée de cassures qui accolent différents segments de manière combinatoire, aboutissant finalement aux gènes codant pour les chaînes des anticorps. Le processus sous-jacent à ces phénomènes de cassures/fusions de segments de gènes s’appelle la recombinaison V(D)J (voir figure).
Les cassures d’ADN au niveau des divers segments sont catalysées par un complexe protéique, appelé complexe RAG, constitué de deux sous-unités : RAG1 et RAG2. Le modèle actuellement en vogue est que la transcription en ARN favorise le recrutement de RAG2 en premier et l’assemblage du complexe permettant à la sous-unité RAG1 (dotée d’activité enzymatique) de cliver l’ADN. Le complexe RAG est préférentiellement recruté dans une région appelée “centre de recombinaison”, très enrichi en activité transcriptionnelle et en modifications épigénétiques qui rendent les segments impliqués dans la recombinaison V(D)J aisément accessibles au complexe RAG. Ce concept d’accessibilité, proposé au milieu des années 80, était basé sur l’observation que la transcription de certains segments du locus de la chaîne lourde d’anticorps (locus IgH) coïncidait avec leur recombinaison. Ceci suggérait que la transcription est partie intégrante des mécanismes de régulation qui contrôlent l’accessibilité des différents segments.
Les études ultérieures sur le rôle de la transcription dans l’accessibilité ont abouti à des résultats contradictoires essentiellement dus aux différents systèmes utilisés (in vitro, substrats transgéniques…). Ainsi, on ne sait toujours pas si la transcription des différents segments est absolument nécessaire à leur recombinaison ou si la transcription est elle-même un produit d’autres mécanismes qui génèrent l’accessibilité des segments au complexe RAG. La controverse est compliquée par le fait que, in vivo, la délétion d’un puissant enhancer, contrôlant les événements transcriptionnels et épigénétiques au sein du centre, aboutit à la suppression de la transcription et de la recombinaison.
Les chercheurs ont adopté une approche alternative. Ils ont inséré une séquence particulière en amont de l’enhancer du centre de recombinaison du locus IgH (appelé enhancer Eµ) en laissant intact cet élément. Ils ont montré que la séquence insérée agit comme un "insulateur transcriptionnel", bloquant les interactions entre l’enhancer Eµ et ses cibles et inhibant du coup la transcription dans le centre de recombinaison.
Par une combinaison d’analyses génétiques, fonctionnelles et mécanistiques, notamment par une analyse de la transcription et de la recombinaison au niveau de la cellule unique, les chercheurs ont montré que la recombinaison peut se produire efficacement dans le centre de recombinaison en l’absence de transcription et que l’activité de remodelage de la chromatine, qui dépend également de l’enhancer Eµ, est maintenue. Par ailleurs, on observe que la sous-unité RAG1 peut être recrutée directement dans le centre de recombinaison en l’absence de transcription et de RAG2.
Le modèle proposé est le suivant : l’enhancer Eµ contrôle le recrutement du complexe RAG dans le centre de recombinaison par deux mécanismes imbriqués mais distincts, chacun déterminant quelle sous-unité sera recrutée en premier pour permettre l’assemblage du complexe et l’initiation de la recombinaison V(D)J. Ainsi, la transcription favoriserait le recrutement de RAG2 et le remodelage de la chromatine celui de RAG1.
Ces résultats pourraient mettre fin à une controverse qui dure depuis des décennies et ouvrent la voie à l’identification plus fine des motifs qui sont responsables du contrôle de la transcription et de la recombinaison par les enhancers impliqués dans les lymphocytes B et T. Ils peuvent apporter un éclairage nouveau dans le cas des translocations chromosomiques associées à la recombinaison V(D)J (également catalysées par le complexe RAG), pouvant aboutir à des leucémies ou lymphomes, mais aussi parce que le complexe RAG est maintenant reconnu comme facteur d’instabilité au niveau du génome entier des lymphocytes B et T. Plus généralement, ces mécanismes de recrutement différents des sous-unités du complexe RAG pourraient aider à mieux comprendre le rôle de ce complexe dans l’instabilité génomique.
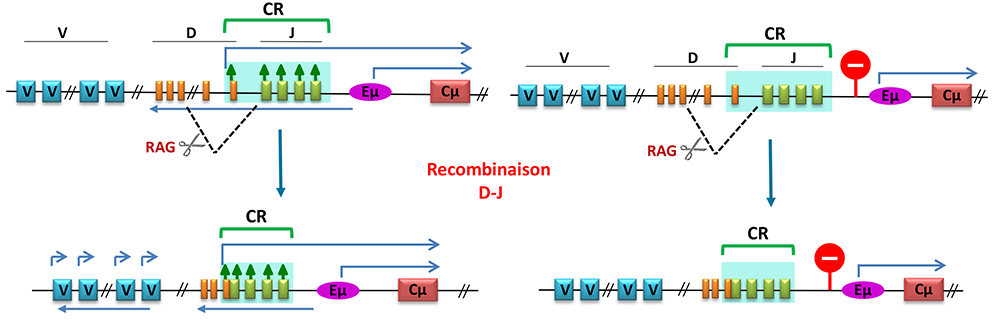
Figure : Schéma du locus IgH murin. Le locus est divisé en deux régions : variable et constante. La recombinaison V(D)J cible la région variable contenant différents groupes de segments : V (variable), D (diversité) et J (jonction). Dans le locus IgH, la recombinaison V(D)J se fait en deux étapes catalysées par le complexe RAG (ciseaux) : un des segments D recombine avec un des segments J (recombinaison D-J), ensuite un des segments V recombine avec le segment DJ pré-formé (recombinaison V-DJ non montrée dans le schéma). Le centre de recombinaison (CR) comprend les segments J et le dernier segment D. L’enhancer Eµ (ovale violet) est situé juste en aval du centre de recombinaison. Les flèches bleues indiquent différents transcrits "sens et anti-sens". Les flèches vertes verticales indiquent des modifications épigénétiques associées à la transcription dans le centre de recombinaison. Dans la partie droite de la figure résumant l'expérience, le signal stop représente l’insulateur inséré entre l’enhancer Eµ et le centre de recombinaison. Cette insertion inhibe la transcription sens et anti-sens qui traverse les domaines D et J ainsi que les modifications épigénétiques associées à la transcription, mais pas la recombinaison D-J.
Pour en savoir plus :
Recombination may occur in the absence of transcription in the immunoglobulin heavy chain recombination centre.
Oudinet C, Braikia FZ, Dauba A, Khamlichi AA.
Nucleic Acids Res. 2020 Feb 22. pii: gkaa108. doi: 10.1093/nar/gkaa108. [Epub ahead of print]
Contact
Laboratoire
Institut de pharmacologie et de biologie structurale (IPBS) - (CNRS/Université Paul Sabatier)
205 Route de Narbonne – BP 64182
31077 Toulouse Cedex 4