Traduction du Virus de la Paralysie du Cricket : le piratage du ribosome d’insectes
Des chercheurs de l’Institut de biologie moléculaire et cellulaire ont élucidé les mécanismes moléculaires permettant au Virus de la Paralysie du Cricket de pirater la machinerie de synthèse des protéines de l’insecte. C’est ainsi que le virus force la cellule infectée à fabriquer ses protéines, qui seront ensuite assemblées pour former de nouvelles particules virales. Cette étude qui ouvre des perspectives pour lutter contre les virus qui s’attaquent aux insectes polinisateurs comme les abeilles, a été publiée le 18 juillet 2017 dans la revue Nucleic Acids Research.
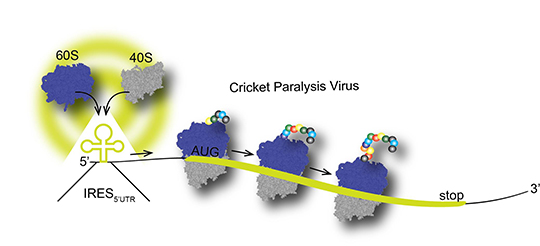
Les virus sont composés d’un acide nucléique (ARN ou ADN), support de leur information génétique, ainsi que de protéines permettant de l’empaqueter dans une particule virale. Les protéines sont synthétisées par une machinerie macromoléculaire complexe appelée ribosome. Le ribosome est assisté par de nombreux facteurs cellulaires pour trouver le site de démarrage de la synthèse des protéines. Cependant, les virus ne possèdent ni ribosomes, ni les facteurs auxiliaires, pour synthétiser leurs propres protéines. Lorsque le virus pénètre dans la cellule hôte, il est obligé de détourner les ribosomes de la cellule pour synthétiser ses propres protéines virales.
Les chercheurs des laboratoires « Architecture et Réactivité de l’ARN » et « Réponse Immunitaire et Développement chez les Insectes » à l’Institut de Biologie Moléculaire et Cellulaire, ont décortiqué les mécanismes moléculaires mis en place par le Virus de la Paralysie du Cricket (CrPV) pour pirater les ribosomes des cellules d’insectes, en l’occurrence la drosophile dans cette étude. Ils ont découvert comment l’ARN viral se replie pour former des structures qui sont capables d’attirer les ribosomes d’insectes. Ces structures particulières sont appelées IRES (Internal Ribosome Entry Site), et l’une d’entre elles, située au milieu de l’ARN viral de CrPV, avait déjà été décrite et souvent étudiée.
Au cours de cette étude les chercheurs ont élucidé au niveau moléculaire le fonctionnement d’une autre séquence IRES localisée en 5’ de l’ARN viral. En effet, grâce à la structure qu’adopte cette IRES, le virus mime la structure de certains facteurs auxiliaires dont il peut dès lors se passer pour attirer le ribosome de drosophile et permettre une synthèse rapide et efficace des protéines virales. De manière intéressante, l’IRES est utilisée par le virus tout au début du cycle infectieux, dans les étapes précoces de l’infection par le virus. Cette particularité ouvre la voie vers des moyens de lutte contre l’infection virale car cette IRES est une cible de choix pour annihiler la propagation du virus dès son entrée dans la cellule. Or le CrPV fait partie d’une grande famille de virus qui s’attaquent non seulement à la drosophile mais aussi à de nombreux autres insectes, notamment aux insectes polinisateurs comme les abeilles.
En conclusion, cette découverte pourrait avoir d’importantes répercussions sur la protection des insectes contre ces virus, afin de préserver leur présence indispensable à la faune au sein de nos écosystèmes.
En savoir plus
-
The IRES 5’UTR of the dicistrovirus Cricket Paralysis Virus is a type III IRES containing an essential pseudoknot structure.
Gross Lauriane, Vicens Quentin, Einhorn Evelyne, Noireterre Audrey, Schaeffer Laure, Kuhn Lauriane, Imler Jean-Luc, Eriani Gilbert, Meignin Carine, Martin Franck.
Nucleic Acids Res. DOI: https://doi.org/10.1093/nar/gkx622 Published: 18 July 2017
