TFIID, un facteur général de la transcription pas si "général" que ça…
L’expression des gènes dans les noyaux de nos cellules débute par l’assemblage du complexe de pré-initiation (PIC) de la transcription permettant le positionnement de l’ARN polymérase II au niveau des promoteurs. TFIID, composé de la protéine "TATA binding protein" (TBP) et de 13 protéines associées, reconnaît les séquences promotrices du gène et initie l’assemblage du PIC. Les chercheurs ont étudié un type cellulaire unique dans lequel l’initiation de la transcription ne dépend pas de TFIID: l’ovocyte. Publiés dans la revue Nature Communications, ces travaux mettent en évidence un nouveau complexe qui remplace TFIID/TBP responsable de l’initiation de la transcription au cours de la croissance ovocytaire.
L’étude de la régulation de l’expression des gènes est une question majeure qui a des implications dans la compréhension des pathologies humaines. Chez les eucaryotes, trois ARN polymérases (Pol) sont responsables de la transcription du génome nucléaire. En particulier, l’ARN Pol II transcrit les gènes codant pour les protéines. La transcription par l’ARN Pol II nécessite le recrutement du complexe de pré-initiation de la transcription (PIC). Sur les séquences promotrices. Le PIC est composé de l’ARN Pol II et de plusieurs complexes protéiques appelés facteurs généraux de la transcription (GTF). TFIID est un GTF conservé au cours de l’évolution et c’est le premier GTF à reconnaître les séquences promotrices et à initier l’assemblage du PIC aboutissant au recrutement de l’ARN Pol II et à l’initiation de la transcription. TFIID est composé de la "TATA-binding protein" (TBP) et de 13 TBP-associated factors (TAFs). Le modèle traditionnel suggérant que la transcription est toujours régulée par les mêmes complexes protéiques a été remis en cause ces dernières années par la découverte de complexes TFIID spécifiques à certains types cellulaires contenant des protéines TAF homologues spécialisées et par l’observation que certains TAFs ne sont pas nécessaires à la transcription dans certains types cellulaires. De plus, trois protéines homologues à TBP ont été identifiées chez les métazoaires dont TBPL2/TBP2/TRF3, spécifique des vertébrés.
Les femmes naissent avec un nombre fixé de follicules primordiaux contenant chacun un ovocyte unique bloqué en prophase de la première division. Au cours de la folliculogenèse, un programme de transcription spécialisé se met en place, permettant la croissance de l’ovocyte par l’accumulation de protéines et d’ARNm. À la fin de cette étape, la transcription cesse et ne reprend qu’après la fécondation : autrement dit, tous les évènements compris entre ces deux évènements dépendent non pas de la transcription active des gènes mais des transcrits et des protéines accumulés.
De manière remarquable, au cours de la croissance ovocytaire, la protéine TBP est absente et remplacée par TBPL2. TBPL2 a un rôle fondamental puisque qu’en son absence, les souris femelles sont stériles et la transcription par l’ARN Pol II est bloquée. Le but de l’étude codirigée par László Tora et Stéphane Vincent était de comprendre comment TBPL2 se substitue à TBP pour l’initiation de la transcription dans ce contexte cellulaire unique en utilisant la souris comme modèle puisqu’il n’existe pas de modèle cellulaire permettant de reproduire l’ovogenèse à grande échelle.
Les chercheurs sont parvenus à isoler les protéines associées à TBPL2 dans les ovocytes. Contrairement à TBP qui est associé aux TAFs, TBPL2 n’interagit pas avec les TAFs mais interagit fortement avec le GTF TFIIA. L’absence de problème de croissance ovocytaire chez les souris femelles mutantes conditionnelles pour le gène Taf7 codant pour une sous unité de TFIID a confirmé l’absence de rôle de TFIID dans ce processus.
TBPL2 contrôle l’expression de nombreux gènes mais aussi d’une classe d’éléments rétroviraux endogènes qui constituent une source de promoteurs alternatifs dans les ovocytes. De manière surprenante, les chercheurs ont aussi observé une augmentation de l’expression d’un grand nombre de transcrits dans les ovocytes mutants pour Tbpl2 qui pourrait s’expliquer par la dérégulation de la machinerie régulant la stabilité des ARNm.
Dans le cadre d'une collaboration avec une équipe de l'Imperial College (Londres), les chercheurs ont analysé la localisation des sites d’initiation de la transcription et ont montré que TBP et TBPL2 ne reconnaissent pas les promoteurs de la même manière.
Ces données proposent un rôle majeur du complexe TBPL2/TFIIA dans la transition vers la mise en place du transcriptome maternel. De manière remarquable, cette transition semble être le pendant de la transition maternelle zygotique qui a lieu après la fécondation et où l’activation de la transcription du génome du zygote est associée à un changement dans la grammaire d’utilisation des sites d’initiation de la transcription.
L’intérêt de cette étude est avant tout fondamental mais permettra de mieux comprendre les problèmes associés à l’insuffisance ovarienne qui constitue une cause fréquente d’infertilité chez les femmes.
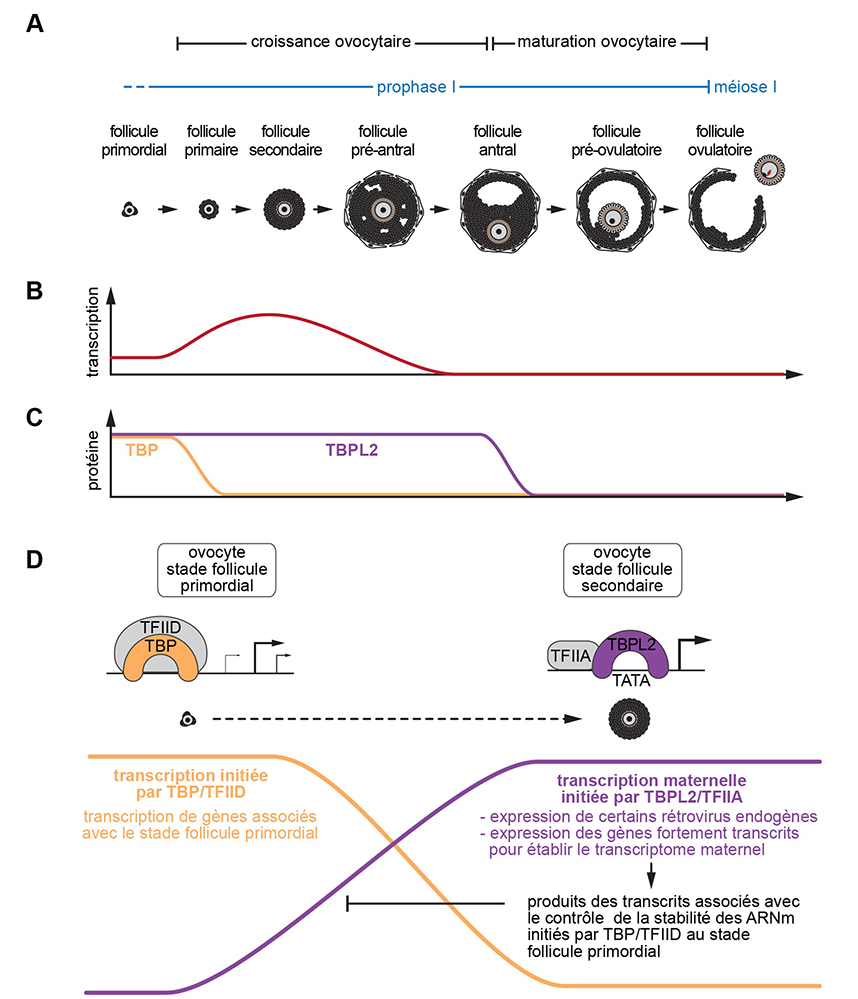
Figure : A) Au cours de la folliculogenèse, la structure des follicules se complexifie et le diamètre des ovocytes bloqués en prophase de la première division méiotique s’accroît jusqu’au stade antral. B) Au cours de cette période, l’activité transcriptionnelle dans les ovocytes augmente et persiste jusqu’à la fin du stade pré-antral. À partir du stade antral, il n’y a plus d’activité transcriptionelle dans les ovocytes. C) Au stade primordial, les protéines TBP et TBPL2 sont présentes dans les ovocytes alors que l’expression de TBPL2 est maintenue jusqu’au stade antral, la protéine TBP n’est plus détectable à partir du stade follicule primaire : autrement dit, l’activité transcriptionnelle pendant la croissance ovocytaire se fait en l’absence de TBP mais en présence de TBPL2. D) Au début de la croissance ovocytaire, la transcription par l’ARN polymérase II (ARN Pol II) est initiée par le complexe TFIID/TBP (orange) à partir de promoteur associé à plusieurs sites potentiels d’initiation de la transcription (TSS). Lorsque la protéine TBP disparaît, l’initiation de la transcription devient sous le contrôle exclusif du complexe TBPL2/TFIIA (violet) via des promoteurs enrichis en séquences proches des boîtes TATA et associés à un TSS majoritaire. TBPL2/TFIIA est responsable de l’initiation importante de la transcription ARN Pol II, y compris de gènes codant pour des protéines associées à la régulation de la stabilité des ARNm déposés précédemment par la transcription ARN Pol II dépendante de TFIID/TBP.
Pour en savoir plus
TBPL2/TFIIA complex establishes the maternal transcriptome through oocyte-specific promoter usage.
Yu C, Cvetesic N, Hisler V, Gupta K, Ye T, Gazdag E, Negroni L, Hajkova P, Berger I, Lenhard B, Müller F, Vincent SD, Tora L.
Nat Commun. 2020 Dec 22;11(1):6439. doi: 10.1038/s41467-020-20239-4.PMID: 33353944
Contact
Laboratoire
Institut de Génétique et de Biologie Moléculaire et Cellulaire (IGBMC) - (CNRS / Inserml / Université de Strasbourg)
1 rue Laurent Fries
67400 Illkirch Graffenstaden (France)