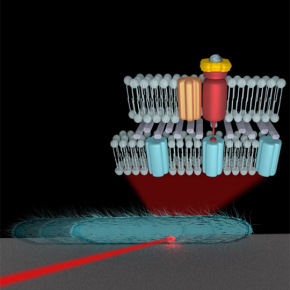
Rotors, stators et embrayages : le mécanisme moléculaire du mouvement des bactéries sur les surfaces dévoilé
Les bactéries se déplacent sur des surfaces par un mécanisme demeuré longtemps mystérieux. Les équipes de Tâm Mignot au Laboratoire de chimie bactérienne et Marcelo Nollmann au Centre de biochimie structurale, montrent pour la première fois que chez Myxococcus xanthus, des complexes protéiques contenant un moteur moléculaire se déplacent selon une trajectoire hélicoïdale parallèle à l’axe de la bactérie. L’immobilisation de ces complexes par des adhésines de la membrane externe entraine de ce fait la propulsion de la bactérie par un mécanisme de vis sans fin.Ces travaux sont publiés dans la revue Nature.
Les bactéries se déplacent sur des surfaces pour former des biofilms et se nourrir d’autres bactéries ou microorganismes. Ce mécanisme, appelé « gliding motility », est resté longtemps mystérieux du fait de l’absence d’organelles extracellulaires visibles. En 2007, Tâm Mignot et ses collaborateurs avaient exploré ce mécanisme pour la première fois en utilisant une approche de microscopie de fluorescence qui s’est révélée très puissante. Elle a permis en particulier de dévoiler que des complexes protéiques associés à la motilité sont assemblés au pôle avant de la bactérie et gardent une position fixe par rapport à la surface de la bactérie en mouvement. Réalisant que les complexes sont en fait en mouvement dans le sens inverse de la bactérie et à la même vitesse, les chercheurs ont postulé que ces complexes propulsaient la motilité via leur attachement à la surface. Ils les ont baptisés « complexes d’adhérence focale bactérienne » par analogie avec les systèmes acto-myosine qui propulsent les cellules eucaryotes.
Au cours des dix dernières années, l’équipe de Tâm Mignot et d’autres laboratoires, ont identifié le complexe de motilité et montré qu’il est effectivement assemblé aux sites d’adhérence focale. Ce complexe contient un moteur moléculaire qui fournit l’énergie de la propulsion en utilisant directement la force proton motrice générée au niveau de la membrane interne. Cependant de nombreuses questions demeuraient non résolues en ce qui concerne le mécanisme de propulsion : quelle est la structure moléculaire du complexe ? Comment les forces sont-elles transmises de la membrane interne à la surface à travers la paroi et la membrane externe ?
Pour répondre à ces questions, l’équipe de Tâm Mignot s’est associée à l’équipe de Marcelo Nöllmann pour développer un nouveau dispositif microfluidique dans lequel la dynamique des complexes peut être suivie avec une haute résolution spatiale et temporelle par microscopie de fluorescence par réflexion totale interne (TIRF). Ceci a permis d’observer que le mouvement hélicoïdal intracellulaire des moteurs processifs provoque la propulsion rotative de la cellule selon un mécanisme de vis sans fin quand ces complexes adhérent à la surface. L’adhérence avec la surface prend place grâce à des interactions transitoires du complexe de membrane interne avec des adhésines de la membrane externe à travers le maillage de la paroi ; un mécanisme qui implique vraisemblablement des bras protéiques rétractiles.
Ce travail résout la base moléculaire du mécanisme de motilité. D’un point de vue général, il révèle l’activité contractile d’une catégorie de moteurs moléculaires qui, s’ils ne permettent la motilité que chez certaines bactéries, sont généralement présents chez les bactéries à Gram négatif où ils ont été impliqués dans des processus tels que l’import de macromolécules dans le cytosol et la division cellulaire.
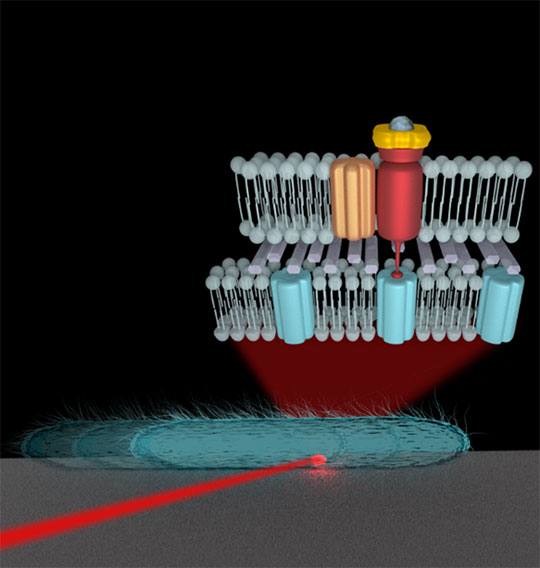
Pour une animation voir :https://naturemicrobiologycommunity.nature.com/users/21217-tam-mignot/videos/12585-myxococcus-xanthus-motility
© Zhanna Santybayeva
En savoir plus
-
The mechanism of force transmission at bacterial focal adhesion complexes.
Nature. 2016 Oct 5. doi: 10.1038/nature20121.
Faure LM, Fiche JB, Espinosa L, Ducret A, Anantharaman V, Luciano J, Lhospice S, Islam ST, Tréguier J, Sotes M, Kuru E, Van Nieuwenhze MS, Brun YV, Théodoly O, L A, Nollmann M, Mignot T.