Ribonucléases enfin capturées en position de maturation sur le ribosome
Chez la bactérie Bacillus subtilis, les étapes finales de maturation des deux ARN ribosomiques de la grande sous-unité du ribosome sont catalysées par des ribonucléases, Mini-III et la RNase M5, capables de cliver des régions double-brin de l’ARN. Les scientifiques ont réussi à visualiser ces ribonucléases sur le point de cliver leur ARNr cible sur la sous-unité 50S du ribosome en développant une stratégie originale de titration utilisant la cryo-microscopie électronique. Ces travaux sont publiés dans la revue Molecular Cell.
Le ribosome, où se réalise la traduction de l’information génétique en protéines, est un énorme complexe formé de deux sous-unités associant ARNs et protéines. Dans la bactérie à Gram-positive, Bacillus subtilis, les étapes finales de maturation des deux ARN ribosomiques de la grande sous-unité 50S du ribosome sont catalysées par des ribonucléases, Mini-III et la RNase M5, capables de cliver des régions double-brin de l’ARN. M5 a été identifié depuis plus de 45 ans, mais sa structure, son mode d’action et le rôle de la protéine ribosomique uL18 dans la réaction de clivage étaient restés complètement inaccessibles. Mini-III est un membre intrigant de la famille des RNase III qui n'a pas le domaine de liaison à l'ARN double brin comme les autres membres de cette famille. Depuis sa découverte il y a une dizaine d’années, la façon dont Mini-III se lie et clive l’ARNr 23S en l'absence de ce domaine était resté mystérieuse.
Les chercheurs ont réussi à visualiser ces deux ribonucléases sur le point de cliver leur ARNr cible sur la sous-unité 50S du ribosome en développant une stratégie originale utilisant la cryo-microscopie électronique. Une des raisons du succès limité de la description des systèmes de maturation provient de la difficulté de capturer les complexes protéine-ARN de faible affinité. Une méthode générale, qui permet de piéger de tels complexes, a été appliquée avec succès à ces deux enzymes de maturation, démontrant la robustesse de la technique. En titrant la particule de plus grande taille, ici la sous-unité 50S du ribosome, avec des quantités croissantes de l’enzyme, beaucoup plus petite, et en visualisant chaque rapport de concentration enzyme/ribosome avec un microscope cryoélectronique, les chercheurs ont pu déterminer le plus grand rapport de concentration enzyme/ribosome qui ne perturbait pas de manière significative le bruit de fond des images et ne présentait donc pas de problème pour leur analyse/traitement. Ils ont ainsi pu résoudre la structure de deux complexes de faible affinité avec un excès d'enzyme de ~200 fois. Cette méthode sera sans doute largement déployée par la communauté scientifique pour capturer des complexes de faible affinité constitués d'un petit et d’un grand composant et facilitera la résolution de la structure de complexes de faible affinité jusqu'ici difficilement accessible.
Ces données fournissent les premières informations structurales sur la maturation de l'ARNr dans les bactéries en révélant comment ces ribonucléases reconnaissent et clivent les ARN de la particule 50S. Mini-III forme un dimère qui lui permet d’occuper les deux sites de clivage de l'ARNr qu’il mature. M5 se lie initialement comme monomère au site de clivage 3’ de son ARNr cible, ce qui impose ensuite un ordre pour cliver chaque brin de l’ARN. Les protéines ribosmiques uL3 et uL18 facilitent le clivage en maintenant une structure spécifique des ARNr reconnues par chacune des RNAses. De plus, uL3 interagit directement avec Mini-III, ceci permettant d’ancrer davantage Mini-III sur la sous-unité 50S.
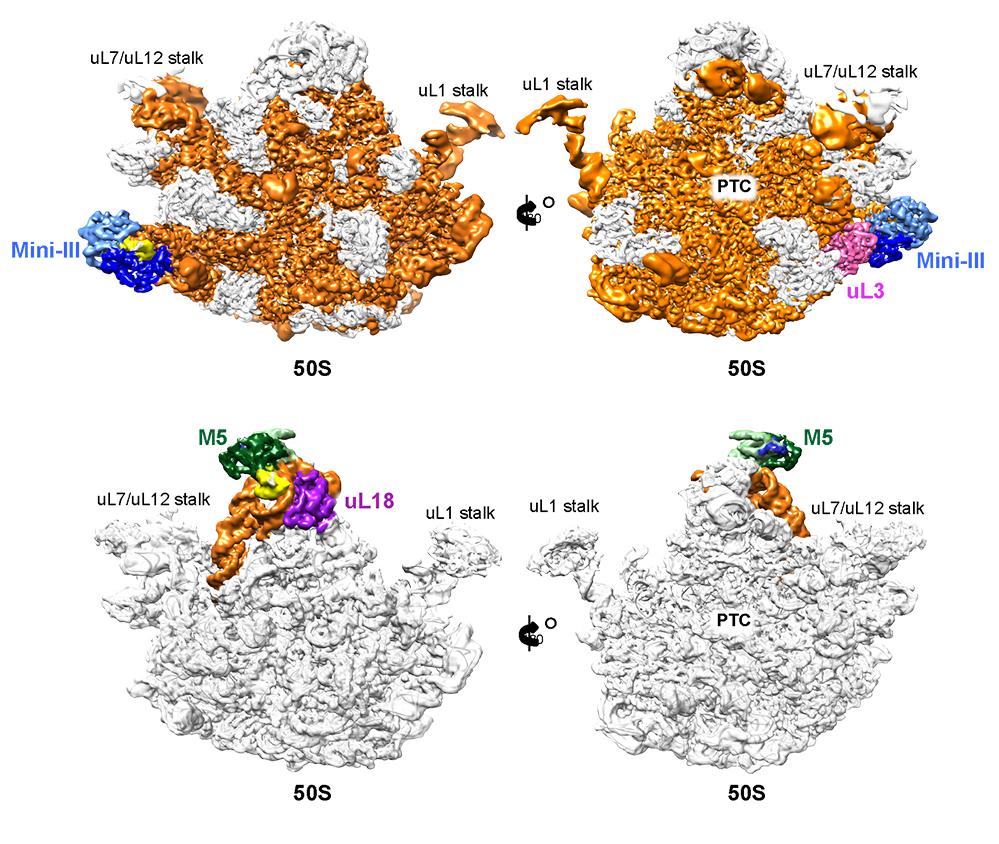
Figure : HAUT :Sous-unité 50S du ribosome de B. subtilis avec la RNase Mini-III (en bleu) piégée sur le point de couper l’ARNr 23S en orange et protéine ribosomique uL3 (en rose) qui permet de maintenir l’ARNr dans une forme reconnue par Mini-III et d’ancrer Mini-III sur la particule 50S. BAS : sous-unité 50S du ribosome de B. subtilis avec la RNase M5 (en vert) piégée sur le point de couper l’ARNr 5S en orange et la protéine ribosomique uL18 (en violet) qui permet de maintenir l’ARNr dans une forme reconnue par Mini-III
Pour en savoir plus:
Structures of B. subtilis maturation RNases captured on 50S ribosome with pre-rRNAs
Oerum S, Dendooven T, Catala M, Gilet L, Dégut C, Trinquier A, Bourguet M, Barraud P, Sarah Cianferani S, Luisi BF, Condon C, Tisné C
Molecular Cell, 28 Sept 2020. DOI: https://doi.org/10.1016/j.molcel.2020.09.008
Contact
Laboratoire
Laboratoire d’Expression génétique microbienne
Institut de biologie physico-chimique (IBPC) - (CNRS/Université de Paris)
13 rue Pierre et Marie Curie, 75005 Paris
