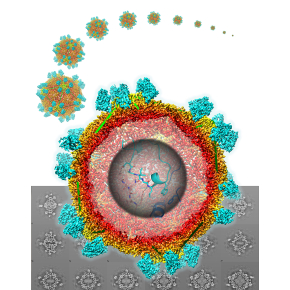
Quand la biologie structurale visualise comment un Nanobody neutralise un virus de plante !
Les scientifiques ont déterminé la structure atomique par cryo-microscopie électronique d’un complexe entre le Grapevine fanleaf virus (GFLV), un pathogène majeur de la vigne responsable de la maladie du court-noué et un fragment d’anticorps neutralisant de camélidés communément appelé Nanobody. La connaissance de cette structure permet de mieux comprendre la façon dont le Nanobody agit pour neutraliser le virus dans la plante et révèle le mécanisme de contournement impliquant des mutations en surface du virus. Ce travail est publié dans la revue Proceedings of the National Academy of Sciences, USA.
La maladie du court-noué de la vigne est d’origine virale. Elle affecte gravement les rendements et la pérennité de nombreux vignobles de par le monde et particulièrement ceux à haute valeur ajoutée. Le principal agent responsable de cette maladie est le Grapevine fanleaf virus (GFLV), un virus transmis spécifiquement de vigne à vigne par un nématode tellurique lors de son alimentation au niveau des racines.
Face à l’absence d’itinéraires techniques pour contrôler cette maladie et de vignes naturellement résistantes au GFLV, des stratégies alternatives de développement de plantes résistantes par des approches biotechnologiques ont été mises en place. L’une d’entre elles consiste à produire des plantes qui expriment de petits peptides dérivés d’anticorps à simple chaîne de camélidés (chameau, lama, alpaca etc.) communément appelés Nanobodies. Que ce soit à partir de plantes modèles de laboratoire ou de vignes cultivées in vitro, l’expression d’un de ces Nanobody, le Nb23, a démontré sa capacité à neutraliser très efficacement et précocement le virus suite à son inoculation.
Afin de mieux comprendre comment le Nb23 neutralise le GFLV in planta, les chercheurs ont déterminé la structure du complexe GFLV-Nb23 à 2.8 Å de résolution par cryo-microscopie électronique (cryo-EM), une méthode d’avant-garde pour l’analyse structurale. Cette étude a permis de montrer que chaque particule virale est recouverte de 60 Nanobodies en condition saturante. Une cartographie précise de l’épitope du virus reconnu par le Nb23 a conduit à identifier tous les acides aminés intervenant dans l’interaction entre le virus et le Nb23. La formation de ce complexe très stable expliquerait la neutralisation précoce et suggère que toutes les étapes du cycle viral où intervient la particule virale peuvent être perturbées. À ce jour, c’est la plus haute résolution en cryo-EM jamais obtenue pour un complexe avec un virus de plante.
Cette étude permet aussi de comprendre comment le GFLV contourne la résistance conférée par le Nb23 qui est produit par la plante, mécanisme qui passe par des mutations modifiant la surface externe de la capside et qui interfèrent avec la reconnaissance du virus par le Nb23. De manière remarquable, la majorité de ces mutations conduisent à des extensions de quelques acides aminés de la protéine de la capside. Ces extensions ne perturbent pas le cycle infectieux du GFLV, mais conduisent à une perte de la capacité du virus à être transmis par les nématodes, empêchant ainsi toute propagation de la maladie de plante à plante. Ce travail permet donc d’établir un lien physique entre la transmission du GFLV par son nématode vecteur et l’épitope de surface reconnu par le Nb23.
Ces résultats mettent en lumière l’importance capitale de l’utilisation de la biologie structurale dans la compréhension des mécanismes moléculaires mis en jeu lors de la résistance conférée à la plante par le Nb23 et constituent un socle qui permettra d’améliorer la robustesse et la durabilité de la résistance dans la perspective de développer des vignes résistantes au GFLV.
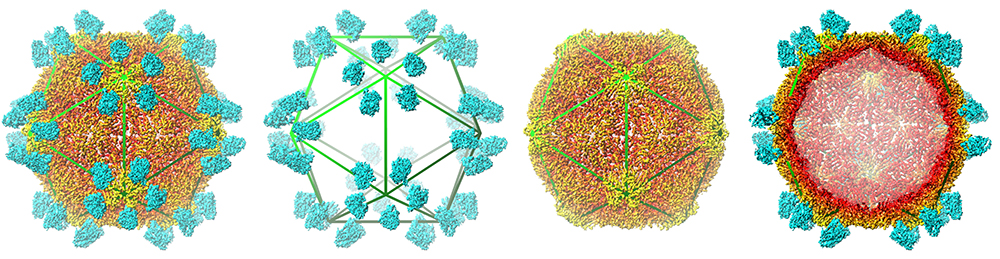
Figure : De gauche vers la droite : structure globale du complexe GFLV-Nb23 avec le Nanobody Nb23 en bleu et la capside en orange, segmentation de la reconstruction 3D avec uniquement la position des Nb23, reconstruction 3D de la capside GFLV seule, coupe transversale à travers la reconstruction 3D du complexe globale. L'organisation en icosaèdre est représentée par un polygone vert superposé.
Pour en savoir plus :
Structural basis of nanobody-recognition of grapevine fanleaf virus and of virus resistance loss.
Orlov I, Hemmer C, Ackerer L, Lorber B, Ghannam A, Poignavent V, Hleibieh K, Sauter C, Schmitt-Keichinger C, Belval L, Hily J-M, Marmonier A, Komar V, Gersch S, Schellenberger P, Bron P, Vigne E, Muyldermans S, Lemaire O, Demangeat G, Ritzenthaler C, . Klaholz BP.
PNAS 19 mai 2020 https://doi.org/10.1073/pnas.1913681117
Contact
Laboratoires
Institut de Génétique et de Biologie Moléculaire et Cellulaire (IGBMC) - (CNRS/université. de Strasbourg/Inserm)
1, rue Laurent Fries, BP 10142, 67404 ILLKIRCH CEDEX
Institut de Biologie Moléculaire des Plantes (CNRS)
12, rue du Général Zimmer, 67084 Strasbourg
INRAE Grand-Est – Colmar
28 rue de Herrlisheim, 68000 Colmar