Maintenir l’hétérochromatine sous silence
L’hétérochromatine, qui réfère aux régions hautement condensées des génomes, contient des séquences d’ADN dont l’expression est inactivée. Dans une étude publiée dans la revue Genome Biology, les scientifiques montrent que l’ADN polymérase epsilon, qui intervient lors de la réplication de l’ADN, assure une fonction cruciale dans le maintien de la structure et de la fonction de l’hétérochromatine.
Dans les noyaux des cellules eucaryotes, les molécules d’ADN sont associées à différentes protéines dont les histones. Cette association forme la chromatine, qui est présente sous deux formes principales appelées euchromatine et hétérochromatine. Comparée à l’euchromatine qui contient la majorité des gènes, l’hétérochromatine est fortement compactée, ne contient que peu de gènes, et est principalement constituée de nombreux éléments transposables et autres séquences répétées dont l’expression est inactivée. L’ADN et les histones de l’hétérochromatine sont associées à des modifications chimiques (marques épigénétiques) caractéristiques. Dans l’hétérochromatine de la plante modèle Arabidopsis thaliana, les cytosines de l’ADN sont fréquemment méthylées, ainsi que les histones H3 au niveau de leurs lysines 9. Ces marques épigénétiques sont généralement nécessaires à l’inactivation de la transcription.
En recherchant des protéines impliquées dans l’inactivation transcriptionnelle de l’hétérochromatine dans le cadre d'une collaboration avec l'Université de Los Angeles Californie et l’institut IPS2 de Paris-Saclay, les chercheurs ont identifié la sous-unité POL2A de l’ADN polymérase epsilon dont l’activité enzymatique permet la synthèse du brin direct lors de la réplication de l’ADN. L’ADN polymérase epsilon est constituée de quatre sous-unités : une grande sous-unité catalytique appelée POL2, et trois sous-unités accessoires (Dpb2, 3 et 4). Le génome d’Arabidopsis contient deux gènes codant la sous-unité catalytique de la polymérase epsilon, POL2A et POL2B, mais seul POL2A est un gène essentiel. En l’absence de protéine POL2A fonctionnelle, la répression transcriptionnelle de l’hétérochromatine est déstabilisée, et l’hétérochromatine, normalement agencée en grosses structures nucléaires, se fragmente. De plus, les résultats démontrent que POL2A inhibe la méthylation de l’ADN et de la lysine 9 de l’histone H3, de telle sorte que l’activation transcriptionnelle de l’hétérochromatine est paradoxalement associée à une augmentation du niveau de ces marques épigénétiques.
D’une manière plus générale, par l’analyse d’autres mutants et l’utilisation de drogues induisant un stress réplicatif, ces travaux révèlent que l'altération de la réplication de l'ADN déclenche systématiquement une augmentation de la méthylation de l'ADN hétérochromatine et déstabilise sa répression transcriptionnelle. Ainsi, cette étude met en lumière le lien fonctionnel étroit existant entre la réplication de l’ADN et le maintien de la structure et de la fonction de l’hétérochromatine.
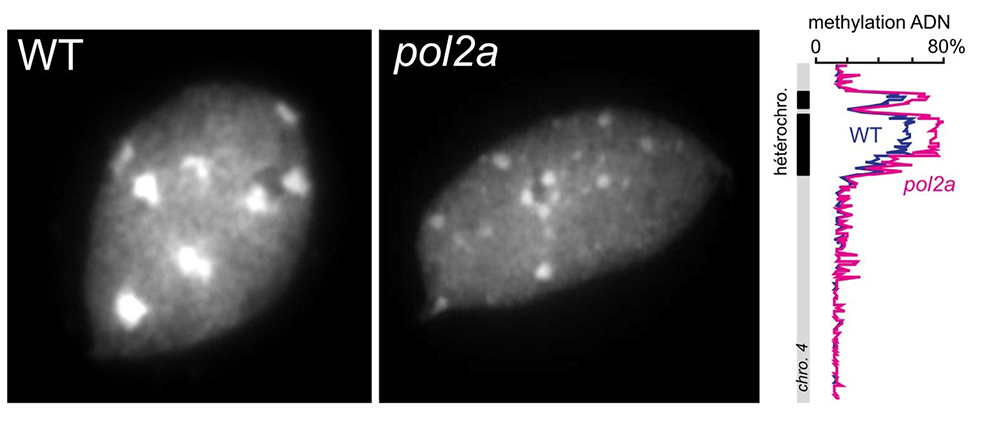
Figure : L’hétérochromatine est altérée dans les plantes portant une mutation du gène POL2A (pol2a). Dans les noyaux des cellules d’une plante d’Arabidopsis thaliana sauvage (WT ; photo de gauche), l’hétérochromatine forme de grandes structures appelées chromocentres hautement fluorescentes après coloration de l’ADN au DAPI. Témoignant d’une structuration anormale de l’hétérochromatine, les chromocentres sont plus nombreux et de taille réduite dans les noyaux de plantes portant une mutation du gène POL2A (pol2a ; photo au centre). Le graphique de droite montre le niveau moyen de méthylation de l’ADN le long du chromosome 4 d’Arabidopsis. Le niveau de méthylation ADN des régions hétérochromatiques (indiquées par des rectangles noirs) est plus important dans le mutant pol2a que dans des plantes sauvages (WT).
Pour en savoir plus :
DNA polymerase epsilon is required for heterochromatin maintenance in Arabidopsis.
Bourguet P, López-González L, Gómez-Zambrano Á, Pélissier T, Hesketh A, Potok ME, Pouch-Pélissier MN, Perez M, Da Ines O, Latrasse D, White CI, Jacobsen SE, Benhamed M, Mathieu O.Genome Biol. 2020 Nov 25;21(1):283. doi: 10.1186/s13059-020-02190-1.
Contact
Laboratoire
Institut Génétique, reproduction et développement (GReD) - (CNRS/ Inserm/Université Clermont Auvergne)
28 place Henri Dunant TSA 50400
63001 Clermont-Ferrand Cedex
