L’intelligence artificielle au service de la médecine : prédire les protéines amyloïdes
L’intelligence artificielle (IA) s’impose aujourd’hui comme une technologie clé dans de nombreux domaines. Dans un article publié dans Alzheimer's & Dementia, des scientifiques démontrent comment l’IA peut prédire la formation des amyloïdes, structures associées à des maladies dégénératives graves. Ces travaux promettent des avancées dans le diagnostic précoce des amyloses héréditaires et autres pathologies liées au vieillissement.
La structure Cross-β : une clé des protéines amyloïdes.
Certaines protéines normalement inoffensives et solubles peuvent, pour des raisons encore mal comprises, commencer à s’agréger en fibrilles amyloïdes insolubles. Ces fibrilles amyloïdes, impliquées dans de nombreuses maladies neurodégénératives comme Alzheimer ou Huntington, partagent une caractéristique structurelle universelle : la structure cross-β. Cette structure, en biologie structurale,est formée par des chaînes polypeptidiques orientées perpendiculairement à l’axe des fibrilles, conférant aux amyloïdes une stabilité et des propriétés communes, indépendamment des séquences spécifiques d'acides aminés des protéines précurseurs. C’est cette organisation qui rend ces agrégats si difficiles à dissocier et si pathogènes dans certains contextes. L’importance grandissante de ces pathologies dans nos sociétés a suscité d’importantes recherches sur les propriétés des séquences d'acides aminés responsables du potentiel d'une protéine à former des amyloïdes. Les méthodes fiables de détection précoce du risque d'amylose faisant encore défaut, les approches informatiques capables de le prédire à partir de données de séquençage du génome des patients à grande échelle sont particulièrement précieuses. Au cours du dernier quart de siècle, plusieurs méthodes informatiques ont été proposées pour prédire l'amyloïdogénicité. Si certains résultats sont prometteurs, il reste encore beaucoup à faire.
L’IA pour prédire les risques et améliorer les diagnostics.
Inspirée par l’importance croissante de l’intelligence artificielle (IA)en biologie structurale et en médecine, les scientifiques ont développé une méthode basée sur l’apprentissage automatique (ML) pour prédire si une séquence protéique donnée peut former des amyloïdes. Cependant, les bases de données existantes mélangeaint souvent différents types d’agrégats, dont certains ne sont pas liés aux amyloïdes « crossβ » associés à la maldie ou formés dans des conditions non pertinentes. Grâce à un travail rigoureux de nettoyage des données, les chercheurs ont créé une base « Cross-Beta DB » spécifique et de haute qualité, leur permettant de développer un prédicteur surpassant les outils existants. Cette innovation, publiée dans la revue Alzheimer § Dementia : The Journal of Alzheimer’s Association, ouvre la voie à des diagnostics précoces et plus fiables des maladies liées aux amyloïdes, en particulier les amyloses héréditaires et neurodégénératives. Le prédicteur Cross-Beta, associé à des données de séquençage génomique, constitue une étape majeure vers des traitements personnalisés et une médecine de précision. Ce projet illustre le potentiel transformateur de l’IA dans les sciences de la vie, où elle contribue à mieux comprendre les mécanismes pathologiques et à anticiper leur progression.
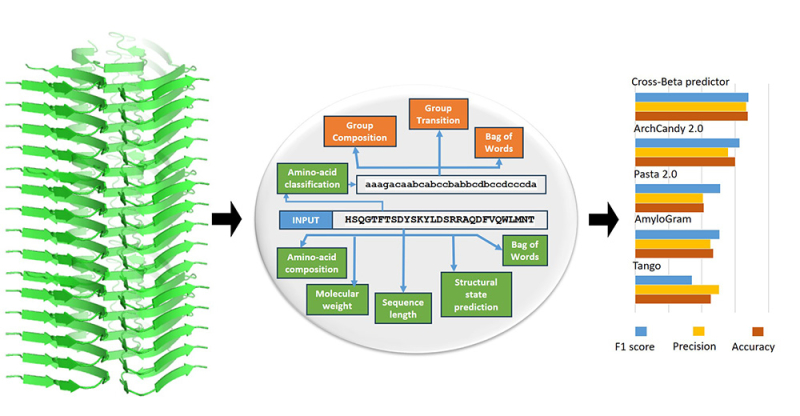
Figure : Ce diagramme illustre les étapes clés de l’étude. À gauche, une structure cross-β typique d'une fibrille amyloïde est représentée. Les amyloïdes cross-β connus ont été compilés dans la nouvelle base de données Cross-Beta DB, accessible via un site web. Cette base de données a ensuite été utilisée pour développer un prédicteur Cross-Beta basé sur ML. Au centre, le diagramme met en évidence les principales caractéristiques extraites des données et utilisées pour entraîner les modèles ML. À droite, le prédicteur Cross-Beta démontre des performances supérieures aux outils de calcul existants. Le prédicteur Cross-Beta peut être utilisé via une interface web.
En savoir plus : Gonay V, Dunne MP, Caceres‐Delpiano J, Kajava AV. Developing machine‐learning‐based amyloidogenicity predictors with Cross‐Beta DB. Alzheimer's Dement. 2024;1‐7. https://doi.org/10.1002/alz.14510
Contact
Laboratoire
Centre de recherche en biologie cellulaire de Montpellier - CRBM (CNRS/Université de Montpellier)
1919 Route de Mende
34293 Montpellier
