Les variations des ARN messagers dépendent de la méthylation de l'ADN
Dans cette étude publiée dans la revue Nucleic Acid Research, les scientifiques montrent qu’il existe une relation causale entre la méthylation de l'ADN et la composition en exons des ARN messagers du gène CD44. La corrélation entre ces deux processus n’a été observée que pour quelques gènes, dont des régulateurs de l’épissage des ARN qui pourraient jouer un rôle de "senseur" traduisant les variations de méthylation de l'ADN en variants d’ARNm pendant le développement de cancers.
La méthylation de l'ADN sur certaines zones régulatrices est une marque épigénétique très largement étudiée dans le contexte du cancer à cause de son rôle sur l’expression des gènes. Mais cette méthylation est aussi présente en dehors de ces éléments et depuis quelques années, plusieurs observations suggèrent un impact au travers d’un autre mécanisme : l’épissage alternatif des ARN. En effet, la plupart des gènes humains sont morcelés en séquences codantes (exons) ou non (introns). Leur expression dépend de la façon dont certains exons sont inclus ou non dans l'ARN messager final (ARNm). On parle alors d’« épissage alternatif » dont les perturbations peuvent contribuer aussi au développement des cancers. Cette étude apporte de nouveaux arguments en faveur d’un impact de la méthylation de l’ADN sur l’épissage alternatif des ARN et surtout montre que sa perturbation dans certains cancers affecte l’homéostasie cellulaire.
Des études précédentes ont décrit des mécanismes par lesquels la méthylation de l'ADN peut influencer l'épissage des ARN. Cependant, la question des poids respectifs des effets directs et indirects de la méthylation de l'ADN n'avait pas encore été clairement abordée. En effet, cette étude montre que l’altération de l'expression des enzymes qui méthylent l'ADN (DNMT) a des effets sur le phénotype épithélial des cellules et, par conséquent, a de multiples effets indirects sur la régulation de l'épissage alternatif.
Les chercheurs ont étudié la relation directeentre la méthylation de l'ADN et la régulation de l'épissage alternatif par le ciblage spécifique à un locus donné, du domaine catalytique DNMT fusionné à la protéine CRISPR/Cas9 inactivée pour la coupure de l'ADN. Comme on peut le voir dans la figure, la méthylation de l'ADN localisée sur des exons variants permet de promouvoir leur inclusion dans les transcrits CD44.
En examinant quatre contextes cellulaires différents, dont un modèle de progression tumorale du cancer du sein et des patients atteints de leucémie aiguë des lymphocytes B, les scientifiques montrent qu'on peut prédire le résultat de l'épissage des transcrits CD44 sur la base de son profil de méthylation de l'ADN. La comparaison des transcriptomes et des méthylomes de cellules d'origines différentes (carcinome du colon et leucémies) a permis d'identifier une petite liste de gènes comme CD44, pour lesquels la régulation de l’épissage alternatif est corrèlée avec des changements de méthylation de l'ADN. Cette liste de gènes contient aussi des facteurs d'épissage dont TRA2B qui est connu pour réguler l'épissage alternatif de CD44. Ces résultats amènent à proposer qu'un nombre limité de gènes puisse être considéré comme des gènes "senseurs" permettant de traduire les niveaux globaux de méthylation de l'ADN en variation d'épissage et en variation d'interactions cellulaires, notamment dans le contexte de la progression tumorale.
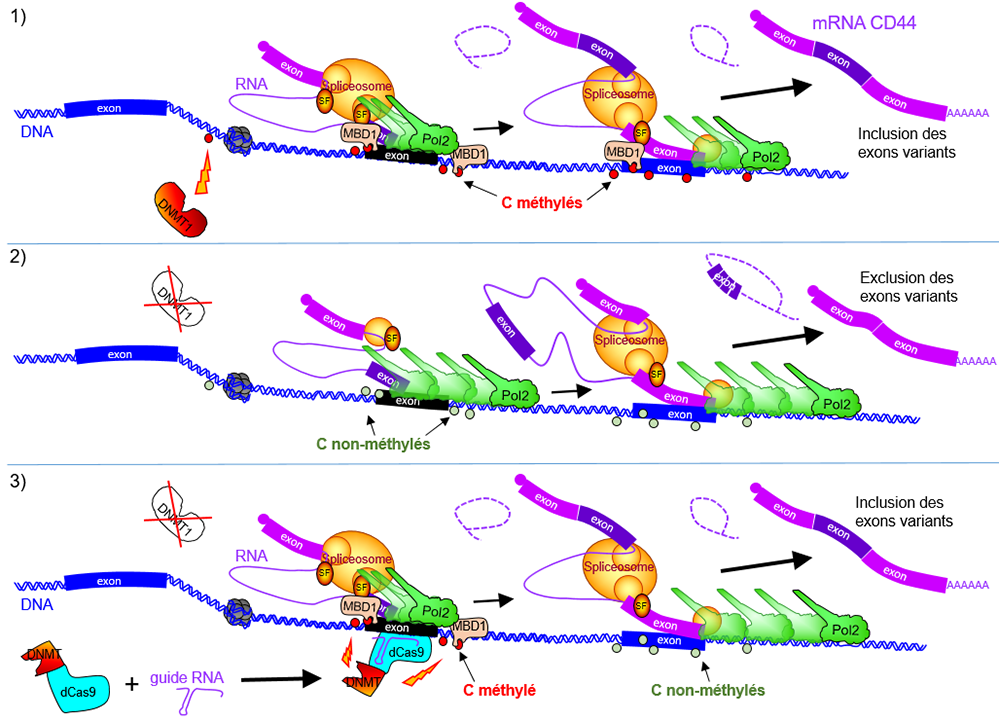
Figure : La méthylation de l’ADN à l'intérieur du gène CD44 favorise l’inclusion des exons variants dans les ARNm. 2) Lorsque la méthylation de l’ADN est perdue, il y a une diminution de la présence sur la chromatine de la protéine MBD1 qui reconnait les Cytosines méthylés (C rouge) et les ARN polymérase II (Pol2) subissent des modifications post-traductionnelles et transcriraient plus rapidement le gène. La machinerie d'épissage (spliceosome) couplée à la transcription reconnaîtrait moins facilement les exons alternatifs. 3) Il est possible d'augmenter localement le niveau de C méthylés en traitant les cellules avec une protéine chimérique portant un domaine catalytique de méthylation de l'ADN fusionné à la protéine CRISPR/Cas9 ciblant les exons variants grâce à des ARN guides complémentaires. Ceci a pour effet de favoriser l'inclusion des exons variants dans les ARNm.
Pour en savoir plus :
CD44 alternative splicing senses intragenic DNA methylation in tumors via direct and indirect mechanisms.
Batsché E, Yi J, Mauger O, Kornobis E, Hopkins B, Hanmer-Lloyd C, Muchardt C.
Nucleic Acids Research. 2021 Jun 4. https://doi.org/10.1093/nar/gkab437
Contact
laboratoire
Adaptation biologique et vieillissement/Institut biologie Paris Seine -IBPS (CNRS/Sorbonne Université)
7 quai St Bernard
75252 Paris cedex 05, France