L’épigénome, la botte secrète des virus géants
Les virus géants, dont la taille et la complexité génétique rivalisent avec les organismes cellulaires, sont omniprésents dans l’environnement. Les scientifiques ont analysé leurs "épigénomes" et montré que leur ADN porte des marques épigénétiques. Les enzymes responsables, aux histoires évolutives complexes, sont parfois associées à d’autres capables de couper l’ADN. Ces systèmes permettraient aux virus géants de dégrader l’ADN des pathogènes présents dans la même cellule, mais aussi de les protéger contre de telles attaques. Cette étude est publiée dans la revue Nature Communications.
Inconnus il y a encore une quinzaine d’années, les virus géants ont une taille qui peut être supérieure à certaines cellules eucaryotes, et des génomes parfois même plus grands que celui de nombre d’organismes cellulaires. L’isolement ces dernières années de virus géants appartenant à différentes familles a mis en lumière la diversité jusqu’alors insoupçonnée de ces virus iconoclastes. Les études de métagénomique environnementale récentes ont confirmé cette diversité et montré qu’ils pourraient jouer un rôle métabolique majeur dans de nombreux écosystèmes. Mais si l’avalanche récente des données génomiques apporte de précieuses informations sur la diversité et l’évolution de ces virus, la fonction de l’immense majorité de leurs gènes reste encore bien mystérieuse.
Au-delà de la séquence d'ADN elle-même, le génome est parfois modifié de manière réversible sans recourir à des mutations. On parle alors d’épigénétique. Cet aspect n’avait jusqu’à présent jamais été étudié chez les virus géants. Or, les modifications épigénétiques jouent un rôle essentiel dans la régulation génique de nombreux organismes cellulaires.
Profitant des dernières avancées technologiques des méthodes de séquençage, les scientifiques ont étudié l’épigénome de virus géants appartenant à différentes familles. Les données de cette étude montrent que la modification de l’ADN par la méthylation est extrêmement répandue, affectant plus de trois quarts des virus étudiés.
Les auteurs ont recherché dans les génomes de ces virus les gènes spécifiant les enzymes, appelées méthyltransférases, responsables de ces modifications. L’histoire évolutive de ces gènes s’est révélée particulièrement complexe puisqu’après les avoir subtilisées aux bactéries, les virus géants ont transféré ces gènes à leur hôte eucaryote et les ont échangées entre eux. Une fois installées dans le génome viral, les methyltransférases sont généralement conservées et induisent probablement un avantage sélectif au virus qui les possède.
L’étude montre que chez certains virus ces enzymes sont couplées à des endonucléases capables de découper l’ADN en morceaux. La co-infection d’une même cellule par plusieurs pathogènes étant courante dans la nature, ce système permettrait au virus d’attaquer un concurrent présent dans la même cellule et de profiter seul de la machinerie cellulaire pour se répliquer. Les méthyltransférases, elles, permettent de protéger l’ADN contre ces coupures. Dans ce cas les virus seraient immunisés contre ces attaques.
Cette première étude ouvre de nouvelles perspectives quant au rôle de l’épigénome dans la dynamique des populations des virus géants.
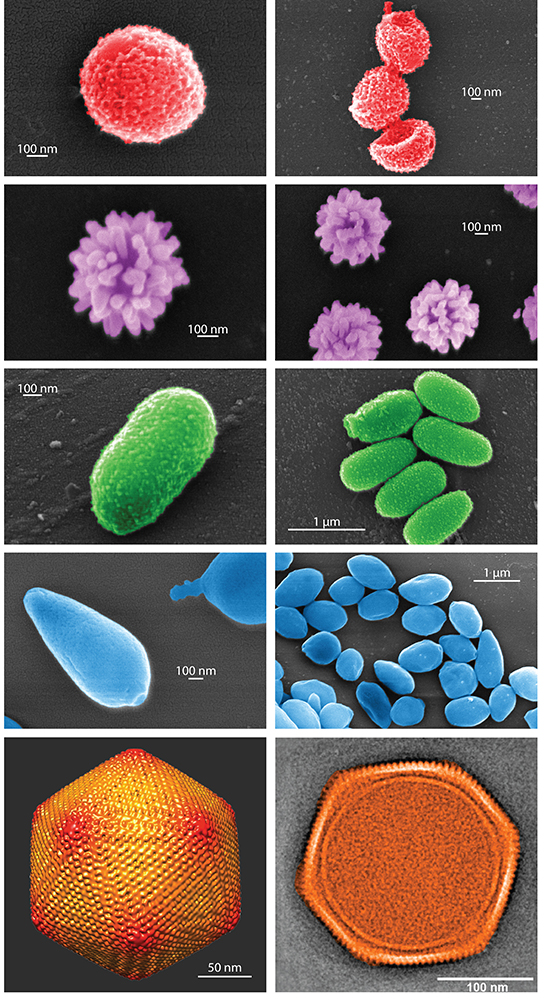
Figure : Aperçu des virus géants dont le méthylome a été étudié. Images de microscopie électronique à balayage de particules virales de Mollivirus sibericum (en rouge), Mimivirus (en violet), Pithovirus sibericum (en vert) et Pandoravirus salinus (en bleu). En orange est présenté une image en cryo-microscopie électronique (à droite) et une reconstruction 3D (à gauche) d’une particule de Melbournevirus.
Pour en savoir plus :
The DNA methylation landscape of giant viruses
Jeudy S, Rigou S, Alempic JM, Claverie JM, Abergel C, Legendre M
Nature Communications 27 mai 2020. doi.org/10.1038/s41467-020-16414-2
Contact
Laboratoires
Laboratoire Information génomique et structurale (IGS) – (CNRS/AMU)
Institut de Microbiologie de la Méditerranée (IMM) – (CNRS/AMU)
Parc Scientifique de Luminy – 163 Avenue de Luminy – Case 934
13288, Marseille cedex 09, France