La variation phénotypique élucidée dans un modèle diploïde
L’équipe de Gianni Liti à l’Institut de recherche sur le cancer et le vieillissement de Nice, en collaboration plusieurs laboratoires en Angleterre, en Suède, et en France, a développé une nouvelle technique facile à mettre en œuvre et très puissante pour décrypter la nature des variations phénotypiques. Ceci a permis de décomposer avec précision les caractères complexes dans le modèle de la levure et d’obtenir ainsi l’analyse la plus aboutie à ce jour chez un organisme diploïde. Cette étude a été publiée le 2 novembre 2016 dans la revue Nature Communications.
Imaginez-vous face à 100 personnes différentes, que verriez-vous ? Ce que vous remarqueriez en premier seraient très certainement les différences observables les plus évidentes, telles que la taille, ou encore le teint de la peau. Cependant, il existe tout autant de variations que vous ne seriez pas en mesure de voir, comme la susceptibilité aux maladies. La plupart de ces traits de caractère sont complexes, c’est-à-dire qu’ils sont déterminés par une multitude de gènes. Ainsi, le fait de comprendre quels sont les gènes et les mécanismes associés qui déterminent un caractère donné, pourrait nous permettre d’expliquer pourquoi certains individus sont plus sensibles que d’autres pour certaines maladies, ce qui constituerait un premier pas vers la médecine personnalisée.
De nombreux progrès ont été réalisés dans l’identification des variations génomiques responsables du développement de diverses maladies, cependant ces études sont encore difficilement réalisables chez l’homme. De ce fait, les chercheurs utilisent généralement des organismes modèles pour mener à bien leurs travaux.
L’équipe de Gianni Liti et ses collaborateurs présentent une nouvelle approche pour étudier les caractères complexes dans des organismes diploïdes, et la mettent en application pour procéder à une analyse détaillée d’un grand nombre de souches diploïdes de Saccharomyces cerevisiae. Cette étude constitue ainsi l’analyse la plus aboutie à ce jour de caractères complexes chez un organisme diploïde.
La stratégie employée permet de travailler à grande échelle et à cout réduit; seulement 170 souches haploïdes ont été séquencées, ce qui permet d’obtenir ensuite la séquence de plus de 7000 souches diploïdes après les avoir croisées entre elles. L’ensemble de ces croisements a été réalisé grâce à un effort commun des laboratoires impliqués, 18 phénotypes différents ont été mesurés pour toutes les souches obtenues. Les différents « Quantitative Trait Loci » (QTLs) ont été déterminés, ainsi que la contribution de chaque composant génétique pour chacun des caractères étudiés. De plus, les mécanismes génétiques responsables du phénomène d’hétérosis (individus dont la valeur d’un phénotype est significativement différente de celle de la moyenne des parents) ont été élucidés. Enfin, l’ensemble de ces ressources a été utilisé pour établir un modèle de prédiction de phénotype, à partir du génome : http://www.nature.com/articles/ncomms11512 .
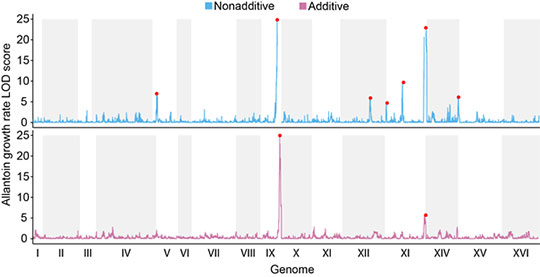
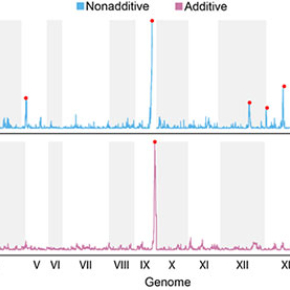
Figure : Les QTL ayant des effets additifs ou non-additifs ont été cartographiés en utilisant un grand ensemble de souches diploïdes. Cet exemple montre la carte QTL déduite des phénotypes de croissance en présence d’allantoïne comme seule source d’azote. Les pics marqués d’un point rouge indiquent les loci qui ont un effet significatif sur le phénotype.
© Johan Hallin
En savoir plus
-
Powerful decomposition of complex traits in a diploid model.
Nat Commun. 2016 Nov 2;7:13311. doi: 10.1038/ncomms13311.
Hallin J, Märtens K, Young AI, Zackrisson M, Salinas F, Parts L, Warringer J, Liti G.