Invasion massive d’éléments génétiques égoïstes dans les gènes de paramécie
Les éléments transposables sont des éléments génétiques mobiles qui ont la capacité de se multiplier au sein du génome de leur hôte en détournant la machinerie cellulaire pour leur propre réplication. Les régions codantes des génomes constituent un no-man’s land pour les parasites intragénomiques, car toute insertion dans ces régions est généralement délétère et donc contre-sélectionnée. Une étude récente publiée dans PLOS Biology montre qu’une classe particulière d’élément transposable a réussi à contourner cette contrainte pour envahir les régions codantes du génome de paramécie.
Les paramécies sont des eucaryotes unicellulaires qui possèdent deux types de noyau : un micronoyau (MIC), diploïde (2n chromosomes), qui est responsable de la transmission de l’information génétique au fil des générations sexuées, mais qui n’est pas exprimé, et un macronoyau (MAC), fortement polyploïde (800n) et où les gènes sont actifs. A chaque génération sexuée, le génome MAC est généré de novo à partir du noyau zygotique (dérivé du MIC). Au cours de ce processus, une fraction importante du génome MIC est éliminée du MAC. En particulier, des travaux précédents avaient montré que le génome MIC de Paramecium tetraurelia contient ~45.000 séquences appelées IES (pour ‘Internal Eliminated Sequences’). Ces IES sont de courtes séquences (longueur médiane ~52 pb), généralement non-codantes, dispersées dans l’ensemble du génome. Ces IES sont excisées très précisément, au nucléotide près, au cours du développement du MAC. La précision de cette excision est essentielle pour pouvoir restaurer des gènes fonctionnels dans le MAC, car 80% de ces IES sont insérées à l’intérieur de régions codantes.
L’origine évolutive de ces éléments génétiques est longtemps restée mystérieuse. Plusieurs indices suggéraient que ces IES pouvaient correspondre à des reliques de transposons. Cependant, la quasi-totalité des IES identifiées chez P. tetraurelia étaient des séquences uniques et ne montrant aucun signe de mobilité récente. Pour comprendre l’origine évolutive de ces éléments, les scientifiques ont séquencé les génomes MIC et MAC de neuf espèces de paramécies. La comparaison de ces génomes a permis de caractériser plusieurs vagues d’insertion d’IES au cours de l’histoire évolutive des paramécies. En particulier, les auteurs ont identifié dans certaines lignées plusieurs milliers d’IES résultant de mobilisations récentes. Ces IES mobiles correspondent à des éléments transposables, le plus souvent non-autonomes (éléments qui peuvent transposer mais ne produisent pas leur propre machinerie de transposition), dont certains ont été acquis par "transfert horizontal" entre espèces. Cette observation constitue la première démonstration directe que des ET sont à l’origine de la prolifération massive des IES dans les gènes de paramécies. En comparant des IES d’âges différents, on peut voir que l’efficacité de leur excision s’améliore au fil du temps, ce qui reflète probablement l'effet d'une pression de sélection pour réduire le fardeau causé par les erreurs d’excision.
La plupart des IES évoluent très rapidement, comme attendu pour des séquences non-fonctionnelles. Les rares exceptions à cette règle correspondent à des IES chevauchant des gènes cellulaires dont l’expression est inactivée par l’excision de l’IES au cours du développement du MAC. Ces exemples révèlent que la machinerie d’excision des IES a pu être recrutée au cours de l’évolution pour contrôler l’expression de ces gènes.
L’évolution des IES et de leur machinerie d’excision offre un parallèle saisissant avec l’évolution des introns des gènes morcelés et leur machinerie d'épissage: dans les deux cas, il s’agit à l’origine d’ET qui ont réussi à coloniser une nouvelle niche dans les régions codantes des génomes, grâce à un mécanisme permettant leur excision avant que le gène soit traduit en protéine. Tout comme les introns, les IES illustrent l’impact des éléments génétiques égoïstes dans l’évolution de la complexité de l’architecture des génomes et des processus d’expression des gènes.
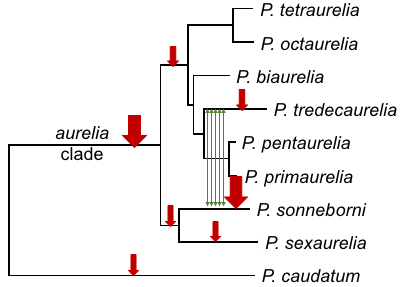
Figure : Le séquençage des génomes de différentes espèces de paramécie a permis de révéler plusieurs vagues d’insertion des IES (flèches rouges) dont deux très massives: l’une ancestrale qui précède la radiation des espèces P. aurelia et l’autre plus récente au sein de la branche P. sonneborni (flèches rouges). Parmi les familles d’IES montrant des signes de mobilité récente chez P. tredecaurelia et P. sonneborni, plusieurs ont subi des transferts horizontaux entre ces deux lignées (flèches vertes).
Pour en savoir plus :
Sellis S, Guérin F, Arnaiz O, Pett W, Lerat E, Boggetto N, Krenek S, Berendonk T, Couloux A, Aury J-M, Labadie K, Malinsky S, Bhullar S, Meyer E, Sperling L, Laurent Duret L, Duharcourt S PLOS Biology 29 juillet 2021. https://doi.org/10.1371/journal.pbio.3001309
Contact
laboratoire
Institut Jacques Monod (Université de Paris, CNRS)
Université Paris Diderot/CNRS - Bâtiment Buffon, 15 rue Hélène Brion,
75205 Paris CEDEX 13
Laboratoire de biométrie et bBiologie évolutive (Université Lyon 1, CNRS)
UCB Lyon 1 - Bât. Grégor Mendel
43 bd du 11 novembre 1918
69622 villeurbanne E cedex