Inhibition allostérique de CRISPR-Cas9
Dans la course aux armements entre les bactériophages et leurs proies, les protéines virales anti-CRISPR inactivent l’immunité bactérienne CRISPR-Cas9. En combinant des approches de biologie structurale, biochimie et biologie cellulaire, les scientifiques décrivent un mécanisme inédit d’inhibition allostérique de CRISPR-Cas9 rendant ce système inapte à reconnaître sa cible. En outre, l’identification d’un système CRISPR-Cas9 naturellement résistant à cet inhibiteur représente l’un des premiers exemples de mécanismes anti-anti-CRISPR. Ce travail a été publié dans la revue Molecular Cell.
Les bactériophages (phages) et leurs proies adaptent réciproquement leurs systèmes d’attaque et de défense pour assurer leur survie. Les bactéries disposent d’un arsenal varié d’outils pour résister aux assauts de leurs prédateurs, dont le système immunitaire CRISPR-Cas9 qui reconnait une région précise du génome viral et le dégrade. En réponse à cette immunité bactérienne, les phages contre-attaquent avec des protéines anti-CRISPR qui inactivent CRISPR-Cas9 et leur permettent d’accomplir leur cycle de vie. L’étonnante diversité des protéines anti-CRISPR, qui ne ressemblent à aucune autre protéine connue, pose de nombreuses questions concernant notamment leur mécanisme d’action et leur impact dans l’évolution de CRISPR-Cas9.
Dans ce contexte, une collaboration internationale et multidisciplinaire entre quatre laboratoires de recherche a permis de caractériser un mécanisme inédit d’inhibition allostérique de CRISPR-Cas9 par la protéine anti-CRISPR AcrIIA6. AcrIIA6 interagit avec une région de Cas9 distincte des sites de liaison de l’ADN envahisseur et des domaines catalytiques. Ce faisant, cet inhibiteur modifie la structure et la dynamique de Cas9, empêchant ainsi la reconnaissance de l’ADN à détruire. De plus, AcrIIA6 a la capacité de séquestrer deux molécules de Cas9 à la fois, ce qui en fait une protéine anti-CRISPR particulièrement efficace pour affaiblir sa proie et assurer la réplication rapide d’une population de phages. Par ailleurs, la caractérisation de ce mécanisme d’inhibition a permis d’identifier un variant naturel de CRISPR-Cas9 résistant à cet inhibiteur. Ce variant, portant des modifications au niveau du site de liaison d’AcrIIA6, illustre la pression de sélection exercée par les protéines anti-CRISPR sur l’évolution et la diversification moléculaire des systèmes CRISPR-Cas9, et représente l’un des premiers exemples de mécanismes anti-anti-CRISPR, encore peu documentés à ce jour.
Cette étude combinant des structures 3D déterminées par cryo-microscopie électronique, des données d’interactions macromoléculaires in vitro, et des tests fonctionnels in vivo, s’inscrit dans le domaine en plein essor de l’étude des protéines anti-CRISPR. Comprendre comment fonctionnent ces protéines est indispensable à notre connaissance des interactions hôte-pathogène, et au développement d’outils biotechnologiques pour le contrôle de l’édition de gènes par les ciseaux moléculaires issus de l’immunité bactérienne CRISPR-Cas9.
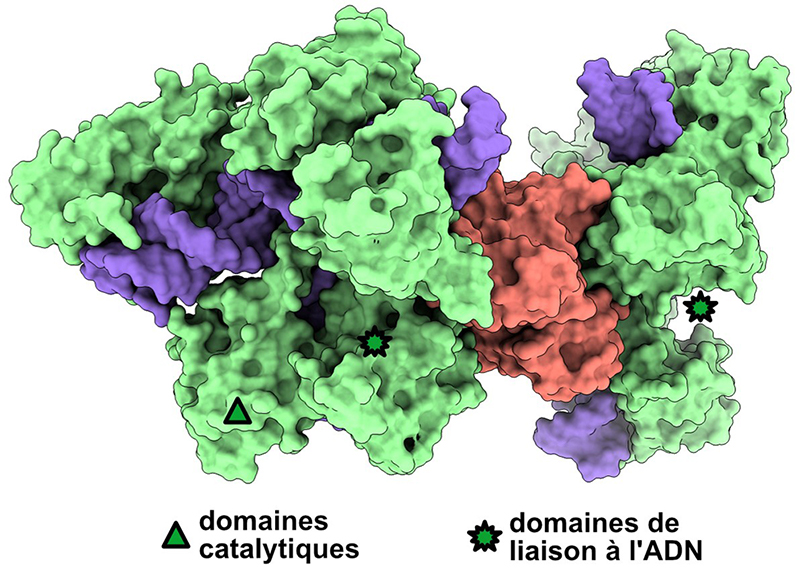
Figure : Représentation de la protéine anti-CRISPR AcrIIA6 (corail) liée à deux molécules de Cas9 (vert : Cas9, acides nucléiques : violet). Le site de liaison allostérique d’AcrIIA6 est distinct des régions de reconnaissance de l’ADN étranger à détruire et des domaines catalytiques.
Pour en savoir plus :
Cas9 allosteric inhibition by the anti-CRISPR protein AcrIIA6.
Fuchsbauer O, Swuec P, Zimberger C, Amigues B, Levesque S, Agudelo D, Duringer A, Chaves-Sanjuan A, Spinelli S, Rousseau GM, Velimirovic M, Bolognesi M, Roussel A, Cambillau C, Moineau S, Doyon Y and Goulet A.
Molecular Cell. October 8, 2019; https://doi.org/10.1016/j.molcel.2019.09.012