Dynamique d’appariement des chromosomes-frères chez Escherichia coli
L’appariement des chromosomes est une étape cruciale à la bonne transmission du matériel génétique qui permet aux paires de chromosomes néosynthétisés de se reconnaître avant d’être ségrégées vers les cellules filles. Dans cette étude, publiée dans la revue Nature Communications, les auteurs s’appuient sur une approche multi-échelle, moléculaire et cellulaire, pour montrer comment la protéine MatP d’Escherichia coli contrôle l’appariement par ses différentes activités.
Les chromosomes, porteurs de l’information génétique, doivent être transmis de manière fidèle lorsque les cellules prolifèrent. Pour cela ils sont dupliqués, puis les copies soeurs sont ségrégées vers les cellules filles. Chez les organismes Eucaryotes, cette étape de ségrégation implique une compaction extrême des chromosomes, qui deviennent alors distinguables en microscopie, un appariement deux à deux des chromosomes frères puis leur transport actif. Chez les bactéries, l’étape de compaction n’existe pas, ce qui a retardé la description de la ségrégation. Nous savons depuis peu que l’étape d’appariement existe mais ne concerne qu’une région particulière des chromosomes. Cette étude révèle que la protéine MatP d’Escherichia coli, connue pour son rôle dans la structuration des chromosomes, agit dans le processus d’appariement de plusieurs manières. Elle montre aussi que son rôle est restreint à cette étape précise du cycle cellulaire.
La plupart des bactéries contiennent un seul chromosome de forme circulaire qui est dupliqué à partir d’une origine unique, définissant une zone de terminaison de la réplication (ter) diamétralement opposée. Cette région ter est aussi la dernière ségrégée vers les cellules filles au cours de la division cellulaire. Elle contient des sites de fixation pour MatP, une petite protéine basique qui agit pour maintenir les régions ter soeur (néosynthétisées) ensemble jusqu’au moment de la division cellulaire, au cours de laquelle elles sont séparées par la translocase d’ADN FtsK.
MatP possède plusieurs activitées. Elle se fixe à l’ADN contenant matS, son site de reconnaissance spécifique, sous la forme de dimères, qui peuvent ensuite former des tetramères. Elle reconnaît la protéine ZapB, médiant ainsi une interaction avec l’appareil de division cellulaire (divisome) assemblé au centre de la cellule sous la forme matS-MatP-ZapB-ZapA-FtsZ. Enfin, elle interagit avec le complexe MukBEF qui possède une activité de condensine et contrôle l’activité de la topoisomérase IV (TopoIV), acteur majeur de la résolution des liens physiques entre chromosomes frère (liens d’intercaténation).
Dans cette étude, les scientifiques ont couplé des approches de biochimie, de biophysique de molécules d’ADN isolées in vitro et de microscopie quantitative de bactéries vivantes pour tenter de comprendre le rôle de MatP dans l’appariement post-réplicatif des région ter du chromosome d’Escherichia coli. Ils ont d’abord montré qu’alors que les formes tétramériques de MatP possèdent deux sites de liaison (un par dimère), ils ne contiennent qu’une molécule d’ADN. Des pontages entre site matS distant peuvent êtres observés in vitro dans différents essais mais sont instables et détruits rapidement par ajout d’ADN non-spécifique. Il est alors fort probable que ce bouclage entre sites matS ne se fasse pas in vivo mais que les formes tétramérique servent pour d’autre activités: interaction transitoire avec de l’ADN non-spécifique et/ou avec d’autres protéines (par exemple ZapB).
La deuxième partie de l’étude, menée lors d’un séjour au Cavendish Laboratory de Cambridge, a permis de montrer que l’on peut déduire l’état d’un locus chromosomique (dupliqué ou non, en appariement ou non) en suivant ses mouvements sur des temps courts à l’aide d’un système permettant le marquage des loci en fluorescence et leur suivi dans des cellules vivantes. Les scientifiques ont ainsi montré que l’effet de MatP, connu pour diminuer la mobilité des loci de la région ter (Coefficient de diffusion et Longueur de confinement plus petits), est restreint à la période d’appariement post-réplicatif. MatP n’a donc que peu de rôle dans la dynamique des chromosomes en dehors de cette période. D’autre part, cette restriction de mobilité dépend au moins de deux activités de MatP génétiquement séparables: l’une dépendant des formes tétramériques et de l'interaction avec ZapB, l’autre de l’activité de la TopoIV. L’appariement des régions ter semble donc faire intervenir deux mécanismes distincts, l’un empêchant leur séparation précoce en inhibant la TopoIV, l’autre assurant leur positionnement dans la zone de division cellulaire où le relâchement de l’appariement peut ensuite se faire de manière contrôlée et couplée à la cytokinèse.
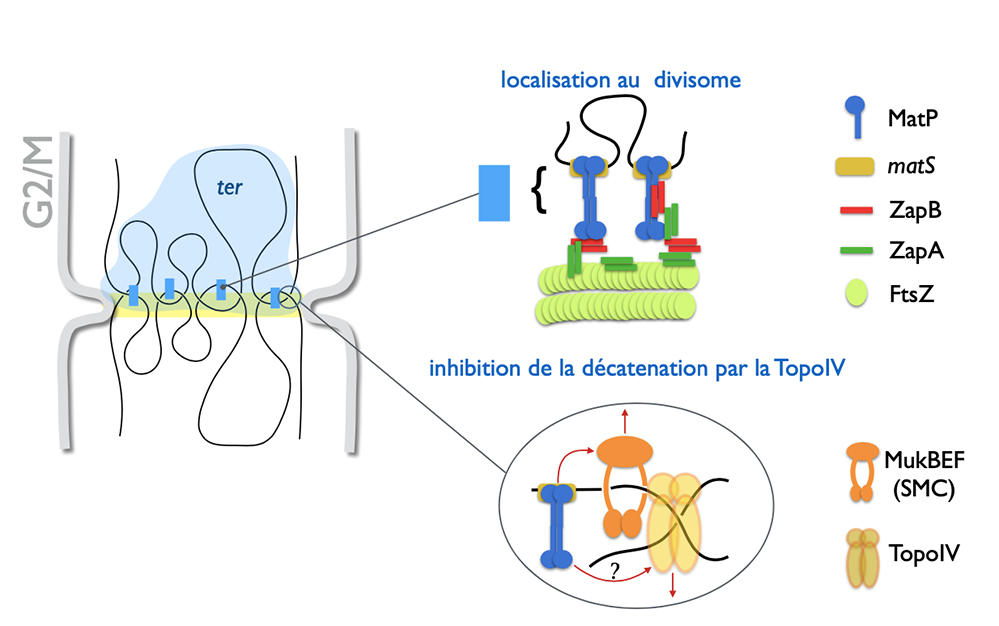
Figure : Le schéma de gauche représente la partie centrale d’une bactérie en train de se diviser (phase G2/M du cycle cellulaire). L'enveloppe cellulaire est en gris. Les parties terminales des chromosomes frères néosynthétisés sont en noir et la zone jaune représente la machine multiprotéique de division cellulaire (le divisome). Les deux mécanismes impliqués dans l’appariement post-réplicatif des régions ter et dépendant de MatP sont schématisés à droite avec leurs légendes. En haut, l’interaction avec ZapB, qui dépend de la formation des tétramères, pourrait se faire par le domaine de fixation à l’ADN libre ou le long d’une structure en “coiled coil” de MatP formée sur les tétramères. En bas, l’exclusion du complexe MukBEF des régions ter (symbolisée par la flèche rouge) pourrait entraîner l'exclusion de la TopoIV par un mécanisme qui reste à décrire. Alternativement, MatP pourrait directement inhiber l’action de la TopoIV (symbolisé par ?).
Pour en savoir plus :
Post-replicative pairing of sister ter regions in Escherichia coli involves multiple activities of MatP
Crozat E, Tardin E, Salhi M, Rousseau P, Lablaine A, Bertoni T, Holcman D, Sclavi B, Cicuta P, Cornet F
Nature communications 30 July 2020. https://doi.org/10.1038/s41467-020-17606-6
Contact
Laboratoire
Laboratoire de Microbiologie et de Génétique Moléculaires (LMGM) / Centre de Biologie Intégrative (CBI) - (CNRS/Université Paul Sabatier)
118 route de Narbonne
31062 TOULOUSE CEDEX 9