D’un cerveau à l’autre : le diable est dans le méthyle!
La fabrication des protéines est réalisée par le ribosome, une macromolécule très complexe décorée par de nombreuses modifications chimiques dont les fonctions biologiques restent énigmatiques. Deux études récentes publiées dans les revues Nucleic Acids Research et EMBO Reports décrivent l’identification de l’enzyme responsable d’une méthylation située au niveau du centre de décodage ou « cerveau » du ribosome. Cette enzyme est importante pour le développement normal du cerveau animal et la marche normale chez la drosophile.
Chez les organismes vivants, la traduction des ARN messagers (ARNm) en protéines est effectuée par le ribosome assisté de nombreux facteurs de traduction et d’ARN de transfert (ARNt). Le ribosome est une nano-machine dont la fabrication chez les organismes eucaryotes est un processus très complexe et hautement coordonné nécessitant plus de 200 protéines d’assemblage et de nombreux ARN non codants. Certains de ces facteurs de biogenèse du ribosome assurent des étapes essentielles de maturation post-transcriptionnelles notamment en décorant l’ARN ribosomique (ARNr) de multiples modifications chimiques (principalement des groupements méthyles). Des défauts de biogenèse du ribosome peuvent ainsi soit être létaux au stade embryonnaire, soit conduire à l’apparition de maladies du sang (principalement des défauts au niveau de la moelle osseuse et regroupées sous le terme de ribosomopathies) ou à des cancers. Si le répertoire de modifications chimiques décorant les ARNr humains est connu depuis plus de 40 ans, l’identification des enzymes les catalysant est encore en cours. Ceci empêche d’étudier le rôle biologique de ces enzymes et de ces modifications post-transcriptionnelles en physiopathologie.
Dans le cadre d'une étude internationale avec l'Université libre de Bruxelles, l'Université de Lausanne et l'institut de biologie moléculaire de Mayence, les scientifiques ont montré que la protéine méthyltransférase METTL5 est essentielle à la formation de la base N6-méthyladenine (m6A) en position 1832 de la petite sous-unité 40S du ribosome humain, une modification qui avait initialement été mise en évidence en 1986. Grâce à des études réalisées chez des archaea (micro-organismes procaryotes dont la machinerie de traduction est assez proche de celle des organismes eucaryotes), ils ont pu identifier que la protéine METTL5 forme un complexe avec la protéine TRMT112, un activateur allostérique de plusieurs méthyltransférases modifiant des facteurs (ARNt, ARNr et protéines) intervenant dans la synthèse protéique.
Cette base modifiée est située au niveau du site de décodage du ribosome qui est important pour l’appariement entre le codon de l’ARNm et l’ARNt spécifique à ce codon et donc pour la fidélité de la traduction. Ce centre fonctionnel du ribosome est communément considéré comme le « cerveau » du ribosome. Les chercheurs ont ensuite inactivé le gène codant pour la protéine METTL5 chez la mouche Drosophila melanogaster et ont observé que les mouches inactivées pour cette enzyme avaient des problèmes de motricité (marche désorientée).
Ces travaux soulignent l’importance d’un petit groupement chimique comme un méthyle décorant le ribosome dans l’expression des gènes et dans le comportement des animaux et font écho à d’autres études très récentes montrant que des mutations inactivant METTL5 provoquent des désordres intellectuels chez l’humain et des microcéphalies et anomalies cranio-faciales chez des animaux.
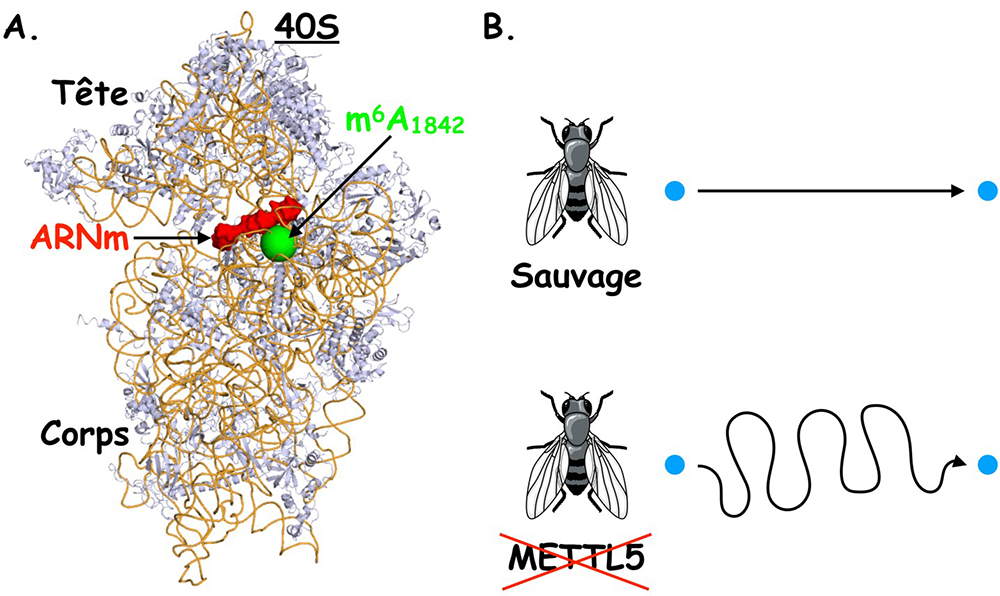
Figure : L’absence de la modification m6A1832 située au niveau du centre de décodage de la petite sous-unité du ribosome provoque des défauts de la marche chez la mouche du vinaigre. (A) Vue de la petite sous-unité du ribosome humain (protéines en bleu et ARNr en orange). L’ARNm en cours de décodage est représenté en rouge. Le nucléotide modifié m6A1832 situé sous l’ARNm dans le centre de décodage est représenté par une sphère verte. (B) L’inactivation du gène METTL5 chez Drosophila melanogaster entraine une marche désorientée.
Pour en savoir plus:
The human 18S rRNA m6A methyltransferase METTL5 is stabilized by TRMT112.
van Tran N, Ernst FGM, Hawley BR, Zorbas C, Ulryck N, Hackert P, Bohnsack KE, Bohnsack MT, Jaffrey SR, Graille M, Lafontaine DLJ.
Nucleic Acids Res. 2019 Sep 5;47(15):7719-7733. doi: 10.1093/nar/gkz619.
The 18S ribosomal RNA m6A methyltransferase Mettl5 is required for normal walking behavior in Drosophila.Leismann J, Spagnuolo M, Pradhan M, Wacheul L, Vu M.A.; Musheev M, Mier P, Andrade-Navarro MA, Graille M.; Niehrs C.; Lafontaine DLJ, Roignant JY
Embo reports, e49443. doi: 10.15252/embr.201949443
Contact
Laboratoire
Laboratoire de Biologie Structurale de la Cellule (BIOC) -(CNRS/ Ecole Polytechnique)
91128 Palaiseau Cedex
