Dialogue intramoléculaire au sein du protéasome humain
Le protéasome est une machinerie protéolytique permettant le recyclage des protéines intracellulaires. Sa dérégulation a été associée à différentes maladies neurodégénératives ainsi qu'à certains cancers. Ce travail, publié dans la revue Nature Communications, révèle un mécanisme de régulation enzymatique à distance entre les sites catalytiques enfouis dans le protéasome et sa surface accessible à ses activateurs. La méthode employée ouvre des perspectives sur l’étude de nombreux autres modulateurs de son activité et sur d’autres complexes multi-protéiques.
Le protéasome est un complexe multiprotéique chargé de dégrader certaines protéines et permettant ainsi de maintenir l’homéostasie protéique. Son activité est également responsable de la concentration intracellulaire de nombreuses cytokines et autres protéines « hub » impliquées dans des processus cellulaires tels la division cellulaire, l’inflammation ou la génération de peptides antigéniques. Elle est donc l’objet d’une régulation très fine dépendant des besoins cellulaires et des perturbations physiologiques ou externes.
Cette activité protéolytique peut être modulée en interagissant avec différents régulateurs, en remplaçant les sous-unités catalytiques ou par modification post-traductionnelle. La littérature dénombre ainsi plus de 300 protéines intéragissant avec le cœur catalytique du protéasome. Les sites catalytiques du protéasome sont cependant enfouis au cœur de ce vaste assemblage multiprotéique (28 sous-unités assemblées sous la forme de 4 anneaux faits de 7 sous-unités différentes), loin de la surface accessible à d’éventuels molécules ou protéines régulatrices. Les mécanismes moléculaires expliquant cette régulation à distance restent ainsi encore mal connus.
Cette étude a permis de mieux décrire ces dialogues intramoléculaires de régulation de l’activité du protéasome, en adaptant une méthodologie innovante à ce gigantesque complexe multiprotéique. Cette technique basée sur l’échange hydrogène deutérium, permet d’estimer l’accessibilité au solvant des différentes régions d’une protéine.
Dès lors, en comparant les données obtenues sur des protéasomes contenant différentes sous-unités catalytiques (protéasomes standard et immunoprotéasomes), les chercheurs ont mis en évidence de fines perturbations intramoléculaires partant de ces sous-unités et se répercutant de proche en proche, jusqu’à la surface du complexe. Ces résultats expliquent comment l’introduction de telle ou telle isoforme au sein du complexe peut favoriser l’interaction avec tel ou tel régulateur spécifique. De plus, la comparaison de ces protéasomes avec ou sans régulateurs a permis de mettre en évidence un mécanisme de régulation "inverse", dans lequel le contact à la surface du protéasome est répercuté jusqu’aux sites catalytiques.
L’utilisation fructueuse de cette méthodologie fondée sur l'échange hydrogène deutérium sur un complexe de la taille du protéasome montre qu’elle pourra sans aucun doute être appliqué à d’autres systèmes supramoléculaires (ribosomes, inflammasomes…).
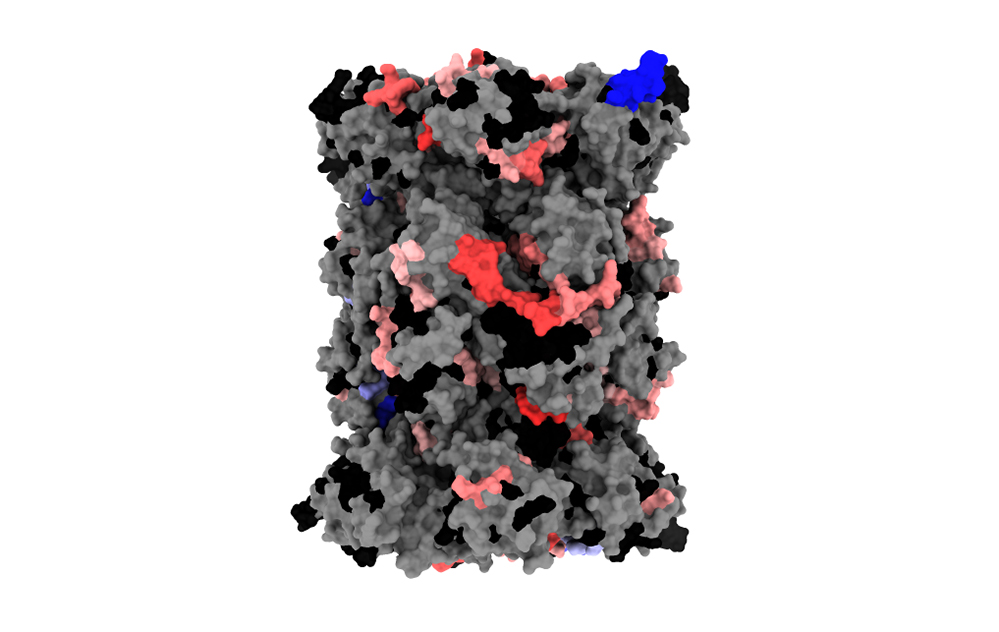
Figure : représentation de la structure 3D du protéasome humain standard (PDB : 5LE5) mettant en évidence les régions étant plus (rouge) ou moins (bleu) flexibles/accessibles après interaction avec l’activateur PA28αβ. Ces résultats d’échange hydrogène-deutérium couplé à la spectrométrie de masse permettent de visualiser les zones affectées lors de la formation du complexe.
Représentation de la structure 3D du protéasome humain standard (PDB : 5LE5)
Audiodescription
Pour en savoir plus :
Conformational maps of human 20S proteasomes reveal PA28- and immuno-dependent inter-ring crosstalks.
Lesne J, Locard-Paulet M, Parra J, Zivković D, Menneteau T, Bousquet MP, Burlet-Schiltz O, Marcoux J.Nat Commun. 2020 Dec 1;11(1):6140. doi: 10.1038/s41467-020-19934-z.
Contact
Laboratoire
Institut de Pharmacologie et de Biologie Structurale (IPBS) - (CNRS/Université Paul Sabatier)
205 route de Narbonne, 31077 Toulouse Toulouse Cedex4
