Dialogue entre plante et virus au front de l'infection
On sait peu de choses sur le dialogue moléculaire entre la plante et un virus envahissant. Dans le cadre d'une collaboration internationale, les scientifiques ont analysé des sites d’infection locale dans des feuilles de colza avec une approche combinatoire à l'échelle du génome pour identifier les réseaux de régulation post-transcriptionnelle guidée par des petits ARN. Les observations révèlent de nouvelles et étonnantes perspectives sur le réseau de régulation à l'interface plante-virus au sein des cellules subissant les premiers stades de l'infection. Ce travail est publié dans la revue Communications Biology.
Les maladies virales causent des pertes importantes de rendement chez les plantes cultivées. Pour développer de nouvelles approches de protection des cultures, il est important de comprendre les mécanismes moléculaires par lesquels les virus propagent leurs génomes infectieux d'une cellule à l'autre et provoquent ainsi une infection systémique. Alors que le mouvement de cellule à cellule du virus est intensément étudiée par imagerie cellulaire, on connait mal le dialogue moléculaire entre le virus envahisseur et le génome de l'hôte. De plus en plus de données suggèrent que les virus végétaux interfèrent avec les voies de régulation médiées par les petits ARN pour modifier l'expression génique dans les tissus infectés. Une telle interaction croisée entre les virus et des plantes a été principalement étudiée au cours de l'infection systémique, alors que l'on en sait toujours très peu sur ce phénomène au front d'infection.
Un réseau de recherche trinational avec l'Allemagne et l'Espagne a été mis en place pour mieux comprendre le dialogue moléculaire entre virus et hôte au front de l'infection. En utilisant le virus de la mosaïque du navet (TuMV) marqué avec la protéine fluorescente GFP (TuMV-GFP) les sites d'infection locale dans les feuilles peuvent être facilement visualisés et excisés. Isolés de cette manière, les sites d'infection de deux cultivars de Brassica napus (dotés de résistance au virus différente) ont été analysés par une approche à l'échelle du génome. L'utilisation de technologies de profilage de pointe et d'outils bioinformatiques a révélé que les petits ARN viraux ainsi que les petits ARN dérivés de gènes codant de l’hôte produisent un jeu de petits ARN induits par le virus qui est utilisé à la fois par le virus et par l'hôte pour réguler l'infection. Ainsi, dans les cellules au front de l'infection, l'hôte utilise de petits ARN endogènes pour cibler le clivage de l'ARN viral et le virus utilise ses propres petits ARN pour cibler le clivage de certains ARN messagers (ARNm) de l'hôte. De manière frappante, il a été découvert que le virus envahissant amène plusieurs gènes de l’hôte à convertir leurs ARN messagers en petits ARN qui s’appelleront « vasiRNAs » (« virus-activated siRNAs »). Dans ce mécanisme, certains gènes de la plante agissent comme des centres de régulation pour produire de petits ARN qui ciblent les ARNm d'autres gènes pour le clivage. Les résultats suggèrent que la susceptibilité des plantes pour les virus peut être expliquée sur la base d'un réseau de régulation complexe et interactif, guidé par des petits ARN et qui fonctionne de manière bidirectionnelle pour contrôler l'expression des génomes viraux et hôtes.
L'infection virale provoque également des changements dans le niveau de transcription des gènes, sans interférence par de petits ARN. Certains de ces gènes sont des gènes liés à la défense antivirale ou sont impliqués dans la régulation des plasmodesmes (PD), les canaux de communication intercellulaire chez les plantes. Par l'activation de la défense antivirale et l'inhibition de la fonction PD, ces gènes pourraient être responsables de l'inhibition de l'infection systémique chez le cultivar le plus résistant.
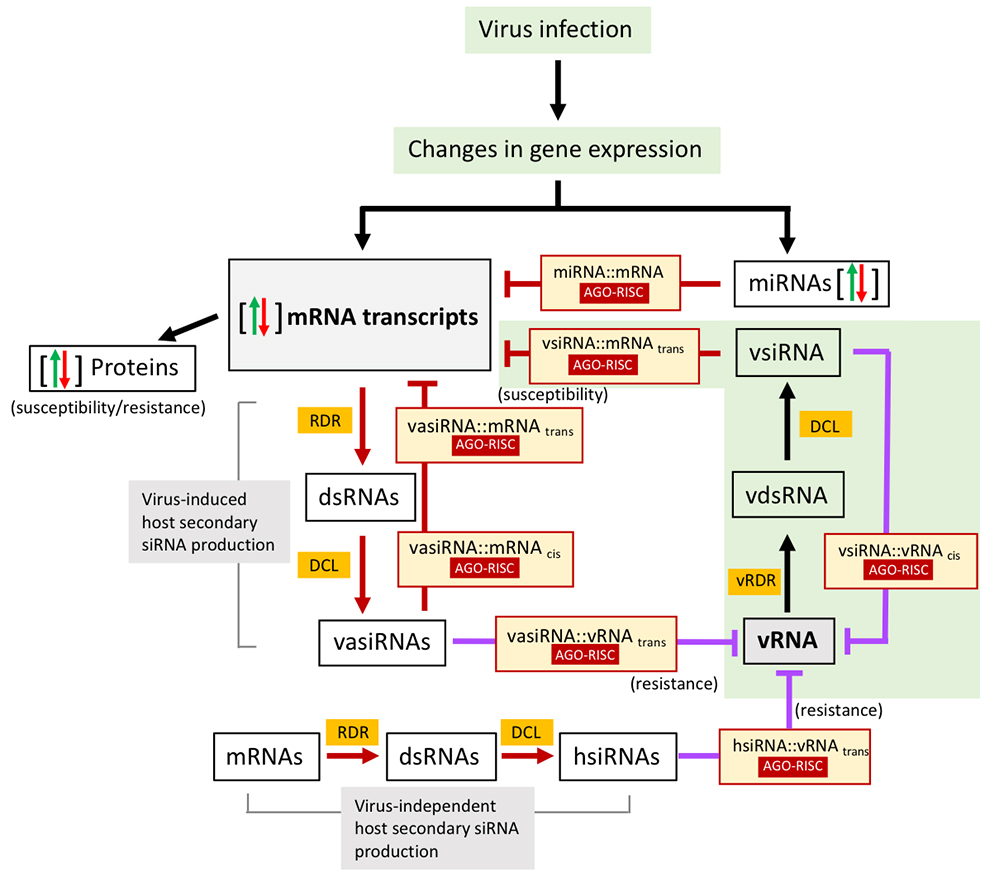
Figure : L'infection virale induit une matrice de régulation complexe d'interactions bidirectionnelles médiées par de petits ARN à l'interface plante-virus. Les perturbations des niveaux de miARN affectent l'amplitude et l'efficacité du clivage médié par les miARN, entraînant des changements dans le niveau de leurs cibles d'ARNm. Le virus provoque l'accumulation de petits ARN interférents activés par le virus (vasiARN) qui sont produits à partir des transcrits d'ARNm de l'hôte par « RNA-dependent RNA polymerase (RDR) », qui convertit l'ARNm en ARN double brin (ARNdb), et des enzmes DICER-LIKE (DCL), qui clivent l'ARNdb en vasiARN. Par la suite, les vasiARN guident les complexes de silençage AGO-RISC pour le clivage des cibles d'ARNm de l'hôte. Les vasiARN agissent soit en cis pour contrôler les transcrits d'ARNm des gènes producteurs de vasiRNA et de leurs homologues, soit en trans pour contrôler les transcrits d'ARNm d'autres gènes cibles. Les siARN dérivés de virus (vsiRNA), qui sont produits par DCL à partir d'ARNd viral (vdsRNA) s'accumulant pendant la réplication de l'ARN viral (ARNv) par l’activité de la RDR viral (vRDR), guident les complexes AGO-RISC pour le clivage d'ARN de l’hôte. De même, l'ARN viral devient une cible accessible des complexes de silencing AGO-RISC dirigés par des vsiRNA, vasiRNA et hsiRNA. Les siRNA dérivés de l'hôte (hsiRNA) sont produits par l'hôte également en l'absence de virus et peuvent contribuer à la résistance basale de l'hôte.
Pour en savoir plus :
Turnip mosaic virus in oilseed rape activates networks of sRNA-mediated interactions between viral and host genomes.
Pitzalis N, Amari K, Graindorge S, Pflieger D, Donaire L, Wassenegger M, Llave C, Heinlein M.
Commun Biol. 2020 Nov 23;3(1):702. doi: 10.1038/s42003-020-01425-y.
Contact
Laboratoire
Institut de biologie moléculaire des plantes (IBMP) - (CNRS)
12, rue du général Zimmer, 67084 Strasbourg, France
