Découverte d’un mécanisme moléculaire permettant le mouvement des bactéries sur des surfaces
Le déplacement des bactéries est un processus essentiel influençant leur capacité à coloniser de nouveaux environnements et à interagir avec d’autres organismes. Dans une étude publiée dans Science Advances, des scientifiques ont découvert le mécanisme moléculaire par lequel la bactérie prédatrice, Myxococcus xanthus, régule l’assemblage des protéines permettant son déplacement sur des surfaces
La motilité bactérienne, indispensable au développement bactérien.
La motilité bactérienne sur surface joue un rôle crucial dans divers processus écologiques et pathogéniques. Dans leur environnement naturel, cette capacité permet aux bactéries de coloniser de nouveaux habitats, de trouver des sources de nutriments et de former des biofilms, qui offrent une protection contre les stress environnementaux. Elle contribue également aux interactions symbiotiques et aux cycles biogéochimiques, influençant la dynamique des écosystèmes. D’un point de vue pathologique, les mouvements des bactéries sur des surfaces sont également un facteur déterminant pour la virulence et la propagation des infections, permettant aux bactéries pathogènes de s'établir et de persister dans les hôtes. Comprendre les mécanismes de la motilité bactérienne sur surface est donc crucial pour le développement de stratégies de gestion des communautés microbiennes et de contrôle des infections.
Décryptage moléculaire du mécanisme de déplacement de Myxococcus xanthus
Chez la bactérie prédatrice du sol, Myxococcus xanthus, les cellules peuvent se déplacer de manière individuelle grâce à l’assemblage d’adhésions focales, structures complexes formées par une machinerie multiprotéique (complexe Agl-Glt) qui connecte le cytosquelette bactérien à la surface sous-jacente permettant à la cellule de se propulser vers l’avant. Ces complexes d’adhésion focale sont assemblés au pôle avant de la cellule et se désassemblent au pôle arrière. Cette régulation spatiale de l’assemblage des adhésions focales est essentielle pour permettre le mouvement directionnel des bactéries, cependant les bases moléculaires et structurales de ce mécanisme de régulation restaient jusqu’à présent inconnues.
Dans cette étude, les scientifiques ont identifié la protéine GltJ comme responsable de la connexion de la machinerie Agl-Glt à la plateforme cytoplasmique contenant le cytosquelette bactérien. Ils ont pu démontrer au niveau structural par résonance magnétique nucléaire (RMN) que GltJ interagit avec les protéines AglZ et MglA-GTP de la plateforme cytoplasmique. Ces interactions ont lieu au niveau du pôle avant de la cellule et induisent un changement structural de GltJ permettant l’exposition de son domaine ZnR. Au niveau cellulaire, les scientifiques ont démontré que cette transition conformationnelle de GltJ permet l’assemblage de l’adhésion focale et le mouvement efficace de la bactérie. De plus, ils ont pu déterminer qu’au niveau du pôle arrière de la cellule, le domaine ZnR exposé de GltJ permet de recruter la protéine MglB. Ce recrutement provoque une hydrolyse rapide du GTP lié à MglA en GDP, entraînant ainsi la dissociation de l’adhésion focale. La protéine GltJ agit donc comme un interrupteur moléculaire permettant la régulation spatiale de l’assemblage des adhésions focales bactériennes ainsi que leur renouvellement lors du mouvement des bactéries.
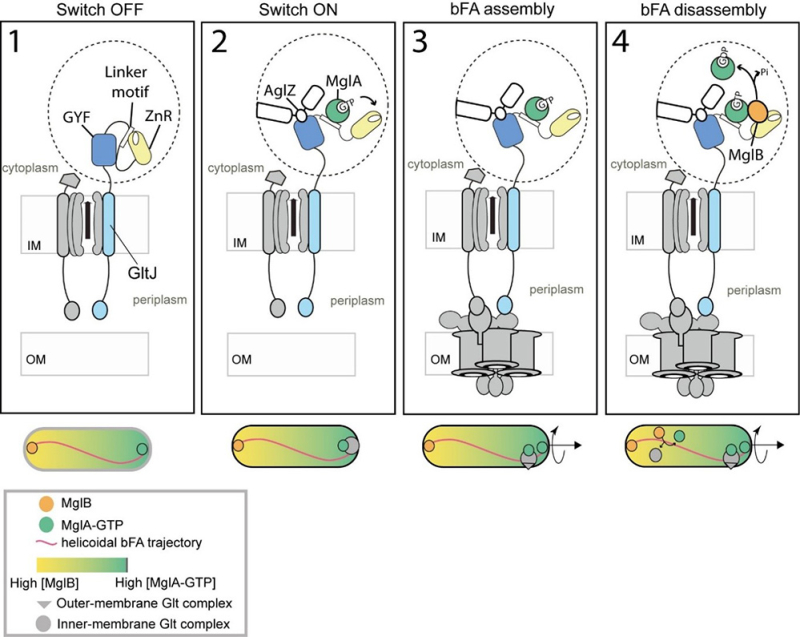
Figure: Assemblage dynamique du complexe Agl-Glt dans les cellules motiles de Myxococcus xanthus. La figure représente le mécanisme proposé d'activation/désactivation des adhésions focales bactériennes par la connexion de la machinerie Agl-Glt aux protéines de la plateforme cytoplasmique (MglA et AglZ). 1. Lorsque le complexe Agl-Glt est inactif et n'est pas connecté à AglZ et MglA, le switch est OFF et le domaine ZnR de la protéine GltJ (en bleu) est dans l'état fermé par l’interaction avec le Linker. 2. Au pôle cellulaire avant, le complexe Agl-Glt devient actif par le recrutement d’AglZ et de MglA-GTP par la protéine GltJ. Le domaine ZnR de GltJ passe alors dans la conformation libre, le switch est ON. 3. Le complexe ainsi activé peut alors interagir avec le complexe de membrane externe afin de former un complexe d’adhésion focale (FAC). La connexion à la surface du complexe Agl-Glt va créer un FAC immobile permettant à la cellule de se propulser sur la surface selon un mouvement hélicoïdal. 4. À l'arrière de la cellule, où la concentration de MglB est plus élevée, MglB est recruté par le domaine ZnR et stimule ainsi l’hydrolyse du GTP par MglA. MglA-GDP ne peut pas se lier au motif Linker et se dissocie ainsi de GltJ entraînant la dissociation du FAC au pôle arrière de la cellule.
Pour en savoir plus :
A molecular switch controls assembly of bacterial focal adhesions
Attia B, My L, Castaing JP, Dinet C, Le Guenno H, Schmidt V, Espinosa L, Anantharaman V, Aravind L, Sebban-Kreuzer C, Nouailler M, Bornet O, Viollier P, Elantak L, Mignot T.
Science Advances, 2024 May 31, DOI : 10.1126/sciadv.adn2789
Contact
Laboratoires
Laboratoire d’Ingénierie des Systèmes Macromoléculaires - LISM (CNRS/Aix-Marseille Université)
31 Chemin Joseph Aiguier
13009 Marseille - France
Laboratoire de chimie bactérienne - LCB (CNRS/Aix-Marseille Université)
31 Chemin Joseph Aiguier
13009 Marseille - France