Décoder l’information spatiale des ARN et de leurs protéines
Il existe un besoin croissant de développer des technologies fournissant des informations quantitatives sur la localisation spatiale des ARN et des protéines à l'échelle de la cellule. Dans cet article, les scientifiques présentent DypFISH, une boîte à outils polyvalente de techniques d’analyse permettant d'interroger les données d’ARN issues de l’imagerie smFISH, en combinaison avec les données d'immunomarquage des protéines. Ces travaux sont publiés dans la revue Cell Reports Methods.
Déterminer la distribution des molécules au sein d'une cellule est essentiel pour définir finement les états cellulaires et comprendre les processus biologiques pour lesquels la traduction d’ARNs en protéines dépend de la localisation intracellulaire. Pour décrypter ce type de phénomènes, les chercheurs ont mis en place DypFISH, une méthode d’analyse de données de microscopie cellulaire de fluorescence (smFISH pour les ARNs et immunofluorescence pour les protéines).
Les progrès réalisés dans le séquençage des ARN messagers (ARNm) ont permis d'identifier des sous-types cellulaires en fonction de la quantité de transcrits dans chaque cellule, cependant différents états cellulaires peuvent également se distinguer par la distribution spatiale subcellulaire des ARN et des protéines.
Afin d’établir des modèles de calcul de localisation et d’interdépendance des distributions spatiales et temporelles des ARNs et des protéines, les auteurs ont exploité le micropatterning cellulaire, c’est-à-dire la culture cellulaire à morphologie contrainte, pour réduire la variabilité intercellulaire. La distribution intracellulaire des molécules a ensuite été caractérisée par des métriques statistiquement rigoureuses, décrivant le clustering (partitionnement de données), la localisation par rapport à des organelles spécifiques ou encore la colocalisation ARNm/protéines. L’outil est disponible sur https://github.com/cbib/dypfish.
Les auteurs ont appliqué DypFISH aux cellules polarisées de fibroblastes de souris micropatternées ainsi qu’à un type cellulaire à morphologie contrainte, les fibres musculaires. Cette méthode a pu mettre en évidence des phénomènes de localisation dans l’espace et dans le temps d’un certain nombre d’ARNs et de protéines liés respectivement aux phénomènes de polarisation et de la maturation des myocytes.
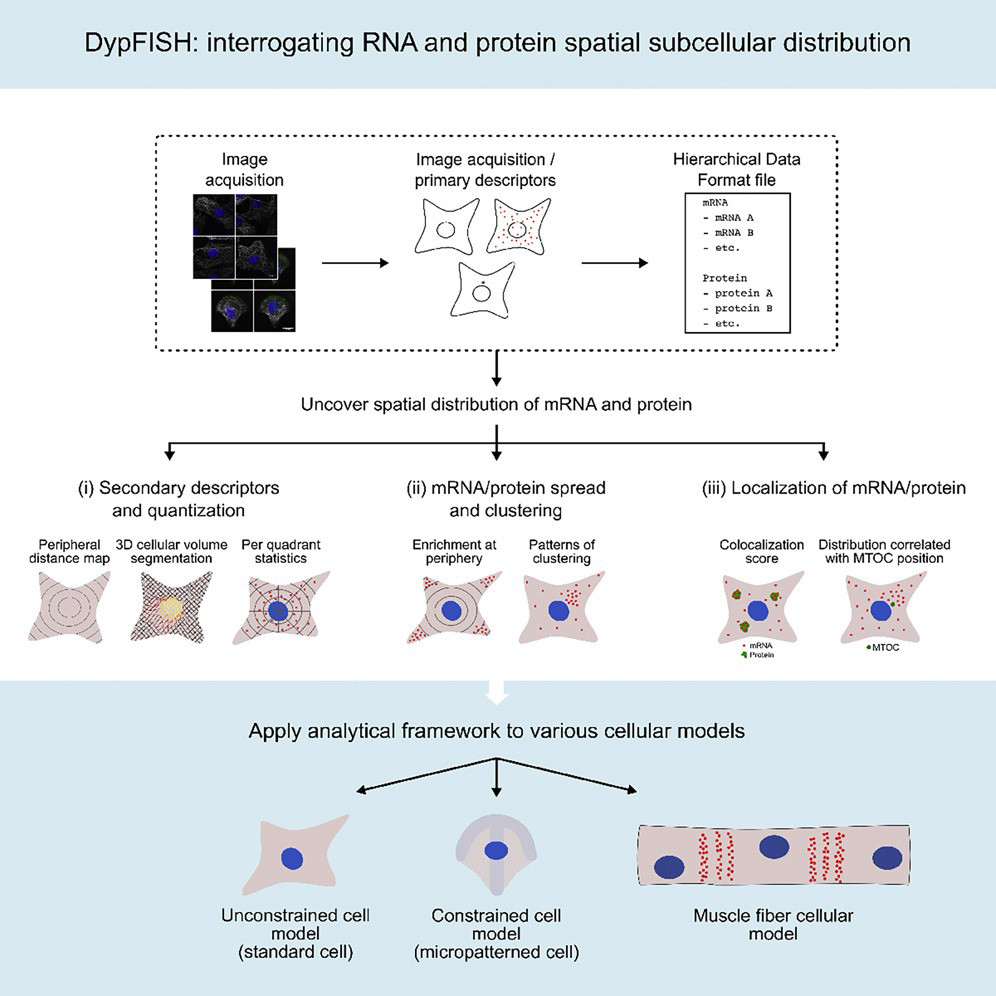
Figure : Les étapes d'analyse des images de microscopie smFISH et IF sont suivies d'analyses statistiques soit (i) pour les images individuelles ou (ii) et (iii) pour des ensembles d'images.
Pour en savoir plus :
Interrogating RNA and protein spatial subcellular distribution in smFISH data with DypFISH
Anca F. Savulescu, Robyn Brackin, Emmanuel Bouilhol, Benjamin Dartigues, Jonathan H. Warrell, Mafalda R. Pimentel, Nicolas Beaume, Isabela C. Fortunato, Stephane Dallongeville, Mikaël Boulle, Hayssam Soueidan, Fabrice Agou, Jan Schmoranzer, Jean-Christophe Olivo-Marin, Claudio A. Franco, Edgar R. Gomes, Macha Nikolski, Musa M. Mhlanga
Cell Reports Methods, 13 septembre 2021. https://doi.org/10.1016/j.crmeth.2021.100068
Contact
laboratoire
Institut de biochimie et génétique cellulaire (CNRS/université de Bordeaux)
Equipe "Computational Biology and Bioinformatics"
1 rue Camille Saint Saëns
CS 61390
33077 Bordeaux cedex
