Créer des protéines de liaison à l’ARN à façon pour réguler l’expression des gènes chloroplastiques
La biologie de synthèse a permis le développement d’outils moléculaires facilitant l’ingénierie des génomes dans les organismes vivants. Toutefois, ces outils sont principalement tournés vers la manipulation des gènes nucléaires et les gènes des organites tels que les mitochondries et les chloroplastes restent encore difficilement accessibles. Dans ce travail, publié dans la revue Nucleic Acids Research, les scientifiques démontrent qu’il est possible d’utiliser des protéines de liaison à l’ARN synthétiques pour contrôler l’expression de gènes-cibles dans les chloroplastes de la plante Arabidopsis thaliana.
Chez les eucaryotes, le contrôle de l’expression des gènes des organites est assuré par des protéines de liaison à l’ARN codées par des gènes nucléaires. Les protéines à domaines répétés en tandem "pentatricopeptide repeat" (PPR) font partie de ces protéines. Elles se lient aux ARN transcrits avec une spécificité de séquence selon un principe modulaire où chaque domaine PPR de 35 acides aminés reconnaît une des 4 bases ribonucléiques grâce à sa séquence. Ce "code PPR" laisse entrevoir la possibilité de concevoir à façon des protéines à domaines PPR ayant la capacité de se lier à des séquences ARN spécifiques dans le but de contrôler l’expression de tel ou tel gène-cible in vivo. Toutefois, les exemples de l’utilisation des protéines PPR naturelles restent rares car leur squelette protéique est difficilement manipulables.
Pour résoudre ce problème, les chercheurs ont conçu des protéines PPR "à façon" qui utilisent un squelette protéique fait de la répétition en tandem d’un domaine PPR synthétique, dont la séquence protéique dérive de la séquence consensus de milliers de domaines PPR trouvés dans la nature. Par des expériences de complémentation fonctionnelle chez la plante Arabidopsis thaliana, les scientifiques prouvent que l’expression de ces protéines PPR synthétiques dans les chloroplastes peut se substituer à la fonction de protéines PPR naturelles en étant capables, in vivo, de reconnaître spécifiquement leurs cibles ARN dans les chloroplastes et de les stabiliser en les protégeant de la dégradation par les ribonucléases. Enfin, ils ont pu démontrer qu’en jouant sur le choix de la séquence ARN ciblée par la protéine, il était possible de contrôler cette dégradation et ainsi de générer des transcrits maturés possédant une nouvelle extrémité 5’ fonctionnelle dans les chloroplastes.
Cette étude révèle pour la première fois la possibilité d’utiliser in vivo des protéines PPR synthétiques comme outil de régulation de la stabilité des transcrits chloroplastiques chez la plante Arabidopsis thaliana. Au-delà, on pourrait imaginer que cet outil s'applique à d'autres organismes utilisant des systèmes comparables.
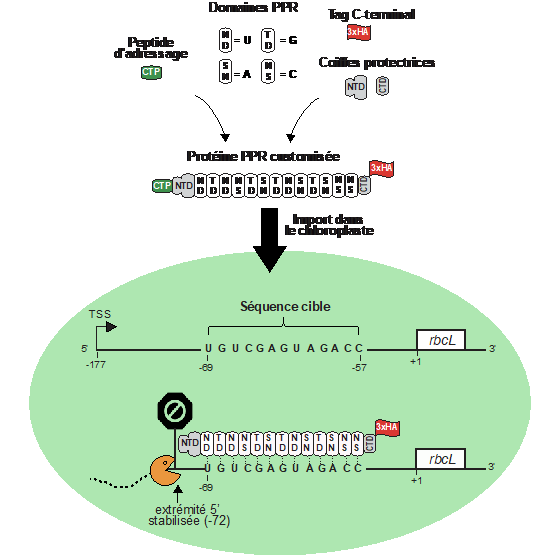
Figure : Contrôle de la stabilité d’un transcrit chloroplastique rbcL grâce à l’utilisation d’une protéine PPR synthétique.
Une protéine de liaison à l’ARN à spécificité de liaison programmable est conçue en concaténant des variants d’un domaine PPR synthétique pour lesquels l’identité de deux acides aminés en positions 5 et 35 de leurs séquences détermine le ribonucléotide reconnu. Les domaines PPR concaténés sont assortis de coiffes protectrices en N- (NTD) et C-terminal (CTD) et d’un peptide d’adressage au chloroplaste (CTP). Une fois exprimée chez la plante, la protéine synthétique est importée dans les chloroplastes où elle liera une séquence ARN cible dans la région 5’-UTR en aval du site de transcription (TSS) du gène rcbL. La liaison de la protéine à l’ARN a pour effet de stabiliser l’ARNm in vivo en protégeant son extrémité 5’ de la dégradation par les ribonucléases chloroplastiques.
Pour en savoir plus :
In vivo stabilization of endogenous chloroplast RNAs by customized artificial pentatricopeptide repeat proteins.
Manavski Nikolay; Mathieu Sébastien; Rojas Margarita; Méteignier Louis-Valentin; Brachmann Andreas; Barkan Alice; Hammani Kamel
Nucleic Acids Research 25 mai 2021 . https://doi.org/10.1093/nar/gkab390
Contact
laboratoire
Institut de biologie moléculaire des plantes (IBMP) (CNRS/Université de Strasbourg)
12, rue du général Zimmer 67084 Strasbourg
