Coopération entre ARN et protéine de la snoRNP U3 et contrôle de la biogénèse des ribosomes
L’assemblage des ARN et protéines constituant les ribosomes (machinerie cellulaire universelle de synthèse des protéines) est très contrôlé. Il implique un grand nombre de facteurs cellulaires, afin que les ribosomes traduisent fidèlement l’information génétique transmise par les ARN messagers. En combinant génétique de la levure et biologie structurale, les scientifiques ont caractérisé un contrôle médié par la mise en place d’une cascade d’interactions entre protéines et ARN au sein du complexe d’assemblage. Ces résultats sont publiés dans la revue Nucleic Acids Research.
Les protéines sont synthétisées par les ribosomes, constitués de plusieurs ARN ribosomiques (ARNr) et de nombreuses protéines qui en forment les deux sous-unités (la petite dite "40S" et la grande dite "60S"). La fidélité de la synthèse des protéines à partir des ARN messagers dépend largement de la qualité des ribosomes. Des erreurs de traduction fréquemment observées dans des cellules tumorales peuvent être extrêmement délétères pour l’organisme. Des travaux récents mettent en évidence l’existence d’un grand nombre de maladies génétiques, liées à des défauts d’assemblage des ribosomes (ribosomopathies). Le haut niveau de qualité requis pour les ribosomes explique l’énergie dépensée par les cellules eucaryotes pour leur biogénèse.
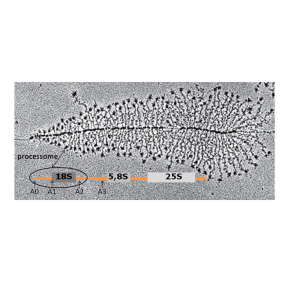
Cette dernière a lieu dans une région du noyau, le nucléole, où sont rassemblés : les gènes codant les ARNr, l'ARN-polymérase I qui les transcrit, les protéines ribosomiques et un très grand nombre de facteurs protéiques et de complexes ARN/protéines (snoRNP) nécessaires à la maturation des ARNr (à partir d’un grand précurseur) et à leur association aux protéines ribosomiques. Les facteurs protéiques s’assemblent en sous-complexes et, dans un premier temps, forment avec les snoRNP et le pré-ARNr un macro-complexe appelé le processome 90S. Celui-ci catalyse les clivages (A0, A1 et A2) du long précurseur, aboutissant à la production de l’ARNr 18S, constituant de la petite sous-unité ribosomique. C'est sur cette sous-unité que se fixe l’ARN messager, tandis que la grande sous-unité prend en charge la traduction de ce messager en protéine. La plupart des snoRNP participent à la modification chimique des ARNr (2’-O méthylations et pseudouridylations). Les ARNr (5,8S et 28S) de la grande sous-unité du ribosome sont produits au sein d’un autre complexe formé dans un second temps. L’ensemble du processus met en œuvre au moins 200 facteurs protéiques (en plus des dizaines de protéines ribosomiques), reflétant l’importance de cette biosynthèse qui doit être rapide et efficace en phase G1 du cycle cellulaire.
Certaines snoRNP n’interviennent pas dans la modification chimique des ARNr mais dans leur clivage. en particulier, le snoARN U3, au centre de cette étude, est requis pour la production de l’ARNr 18S. Les chercheurs ont montré que pour assurer ses fonctions, il doit s’apparier à plusieurs segments du long précurseur des ARNr. Les récents progrès de la cryo-microscopie électronique ont permis de confirmer plusieurs de ces interactions et mis en évidence leur positionnement relatif par rapport aux facteurs protéiques et protéines ribosomiques au sein du processome 90S. Néanmoins, l’assemblage et l’activité du processome implique des transitions structurales, et les structures 3D établies comportent encore des zones non résolues, ne permettant pas d’avoir une compréhension fine de son mécanisme d’action. Ainsi, si le caractère essentiel de la protéine Rrp9/U3-55K associée au snoARN U3 dans la production de l’ARNr 18S était établi, les données structurales ne permettaient pas d’identifier précisément son rôle.
En s'appuyant sur des études génétiques chez Saccharomyces cerevisiae, notamment par mutagénèse dirigée exhaustive des acides aminés de surface du domaine structuré de la protéine Rrp9, les chercheurs ont montré que l’arginine en position 289 est nécessaire à l’efficacité des clivages du pré-ARNr. Par des études d’interactions entre protéines du processome 90S par la méthode double-hybride, ils ont identifié un réseau d’interactions protéine-protéine auquel appartient Rrp9 et identifié les domaines d’interaction. La mutation de l'arginine 289 en alanine affecte l’interaction entre Rrp9 et Rrp36, une protéine également essentielle aux clivages aux sites A1 et A2.
Point important, les scientifiques ont montré un effet synergique négatif de la mutation Rrp9 R289A avec les mutations affaiblissant les appariements entre le pré-ARNr et le snoARN U3. Une analyse des structures 3D des complexes successifs du processome suggère que Rrp36 et Sgd1 en contact avec Rrp9 assurent le bon positionnement de deux très grandes protéines Utp10 et Utp20 formant deux grands arches au sein du processome 90S. Ce positionnement précis serait requis pour la stabilisation des interactions entre le snoARN U3 et le pré-ARNr et par conséquence pour l’efficacité des clivages du pré-ARNr.
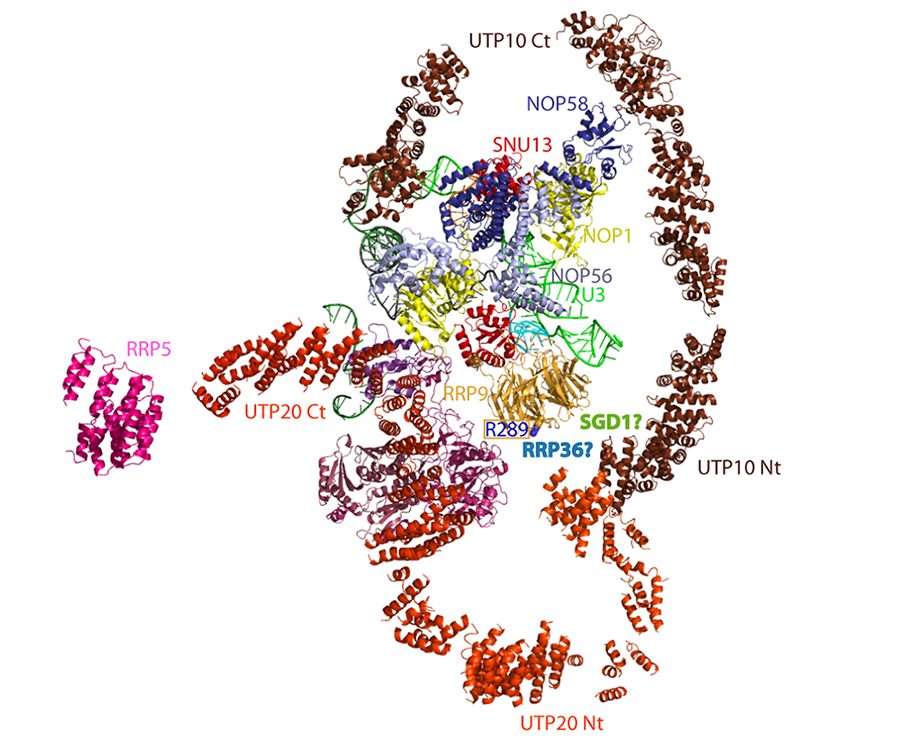
Figure : La protéine Rrp9 de la snoRNP U3 située au coeur des deux arches formées par les protéines UTP10 et UTP20 et les interactions entre le snoARN U3 et le pré-ARNr contribuent de manière synergique à l'efficacité catalytique du processome 90S.
Pour en savoir plus :
Synergistic defects in pre-rRNA processing from mutations in the U3-specific protein Rrp9 and U3 snoRNA
Clerget G, Bourguignon-Igel V, Marmier-Gourrier N, Rolland N, Wacheul L, Manival X, Charron C, Kufel J, Méreau A, Senty-Ségault V, Tollervey D, Lafontaine D, Branlant C, Rederstorff M.
Nucleic Acids Res. 2020 Apr 17; doi: 10.1093/nar/gkaa066
Contact
Laboratoire
Ingénierie Moléculaire et Physiopathologie Articulaire (IMoPA) - (CNRS/Université de Lorraine)
9 Avenue de la Forêt de Haye BP
54505 VANDOEUVRE LES NANCY CEDEX