Comment le métabolisme d’une bactérie contrôle le comportement de la communauté
Dans la nature, de nombreuses bactéries coexistent, formant une « société avancée » reposant sur des interactions. Ces travaux, publiés dans la revue mBio, reposent sur une approche interdisciplinaire et démontrent que la mise en place d’interactions physiques et métaboliques entre deux bactéries implique une petite molécule -AI-2- capable de médier la communication entre des bactéries d’espèces différentes en fonction de leur état métabolique.
Le comportement des bactéries est, en général, abordé au laboratoire par l’étude de systèmes modèles, se développant en culture pure en condition optimale ou dans des conditions très spécifiques de stress. Toutefois, dans la nature, ces conditions idéales n’existent pas et les bactéries coexistent et interagissent physiquement et métaboliquement entre elles. Ces interactions confèrent des propriétés et des capacités nouvelles, non prédictibles par la simple analyse du génome et permettent entre autre aux microorganismes de se développer dans des environnements qui sont peu propices à la vie (absence de substrats, conditions physicochimiques défavorables, etc….). La connaissance des bases moléculaires qui contrôlent la mise en place de ces interactions et donc le comportement de la communauté constitue un apport au décryptage du fonctionnement des écosystèmes mais c'est aussi une source potentielle d’innovation technologique dans le cadre de l’ingénierie écologique en permettant de piloter le comportement d’une communauté microbienne vers un fonctionnement précis.
Dans des environnements anaérobies, la dégradation de la biomasse qui conduit à la production de biogaz fait intervenir différentes espèces ce qui nécessite un couplage et une synchronisation métabolique forte afin d’optimiser cette dégradation en évitant tout effet d’inhibition. L’une des façon d’étudier un tel processus afin d’en décrypter le fonctionnement au niveau moléculaire est de reconstituer des consortia synthétiques, associations de bactéries connues pour intervenir dans ce processus. Un consortium synthétique constitué de deux bactéries i.e. Clostridium acetobutylicum et Desulfovibrio vulgaris, présentes toutes les deux dans la dégradation de la biomasse, a été constitué. Ces deux bactéries sont connues pour l’une (C. acetobutylicum) comme étant responsable de la production d’hydrogène via la fermentation des sucres, pour l’autre (D. vulgaris) comme étant impliquée, en fonction des conditions environnementales, dans la production ou la consommation d’hydrogène.
La mise en place d’une approche interdisciplinaire associant biologistes, chimistes, et génie des procédés, a permis de démontrer que dans des conditions de stress nutritionnel, la mise en place d’interactions physiques et métaboliques entre les deux bactéries reposait sur la production et la détection d’une petite molécule, AI-2, connue pour médier la communication entre des bactéries d’espèces différentes. L’ajout des substrats spécifiques à chacune des bactéries conduit à la disparition de ces interactions et à la production d’une molécule antagoniste de AI-2 qui bloque cette communication. Ceci démontre que les interactions entre bactéries sont finement régulées et contrôlées par le métabolisme de chacune d'entre elles. L’étude d’un autre consortium constitué de Escherichia coli et D. vulgaris souligne le caractère générique de ce mode de communication inter-espèces.
L’une des conséquences de ces interactions et de ce processus de signalisation dans le consortium modèle est une augmentation de la production d’H2 et les chercheurs ont démontré, via l’utilisation d'un substrat marqué, que cela repose sur un échange de carbone entre les deux bactéries.
Ces travaux ouvrent des perspectives prometteuses pour le développement de procédés de production d’H2 "vert" ou décarboné en optimisant des processus microbiens dont ont maitriserait mieux les métabolismes et les interactions entre populations.
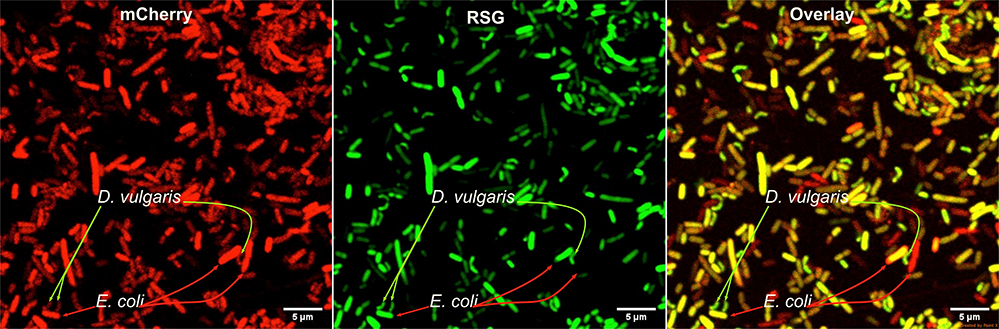
Figure: Sans source de carbone pour se développer, en consortium avec la bactérie E. coli productrice de la molécule AI-2 (mCherry, rouge), D. vulgaris est capable de produire son énergie comme le montre la fluorescence du composé RSG (Redox Sensor Green - vert)
Pour en savoir plus :
Metabolic exchange and energetic coupling between nutritionally stressed bacterial species: role of quorum sensing molecules.
Ranava D, Backes C, Karthikeyan G,Ouari O, Soric A, Guiral M, Cárdenas ML,Giudici-Orticoni MT.
mBio 12:e02758-20. https:// doi.org/10.1128/mBio.02758-20.
Contact
Laboratoire
Laboratoire de Bioénergétique et Ingéniérie des Protéines (BIP) - (CNRS/AMU)
31 chemin Joseph Aiguier 13009 Marseille