Chez la bactérie, l’activation de chaque gène induit la formation de mini domaines à l’intérieur des chromosomes
Dans un article publié dans la revue Nature Structural and Molecular Biology, les scientifiques montrent que les chromosomes bactériens sont constitués d’une succession de petits domaines en trois dimensions représentant chacun un gène ou un opéron (ensemble de gènes liés qui sont régulés ensemble) en activité. Ces domaines peuvent s’associer pour en définir de plus vastes et forment ainsi la base de l’organisation des chromosomes bactériens.
Les génomes bactériens sont organisés au sein du nucléoïde, un compartiment sans membrane où interagissent ADN, ARN et protéines. Cette organisation est modulée par la réplication ou encore la transcription, qui modifient par exemple le surenroulement de la molécule d’ADN. Depuis une vingtaine d’années les chercheurs ont compris que bien que plus petits et contenus dans un compartiment sans membrane les chromosomes bactériens étaient organisés tout aussi hiérarchiquement que les chromosomes des eucaryotes localisés dans le noyau.
Des données génétiques, de biologie cellulaire et de capture de conformation chromosomique (Hi-C), une technique qui permet d’analyser la structure spatiale de la chromatine, ont suggéré une organisation des gènes et des opérons en domaines plus vastes imbriqués les uns dans les autres. Il était admis que les plus petits domaines, appelés des CIDs pour Chromosome Interaction Domains, englobaient plusieurs dizaines de gènes (entre 30 – 300) et étaient séparés par des barrières fixes formées par de longs gènes fortement exprimés. Cependant, les analyses ayant amené à la caractérisation de ces derniers n’étaient pas suffisamment résolutives pour révéler l’organisation d’un gène ou d‘un opéron.
Une résolution dix fois plus importante que les approches classiques
Dans une étude publiée dans la revue Nature Structural and Molecular Biology, les scientifiques ont combiné une approche avec une résolution dix fois plus importante que les approches classiques et des manipulations génétiques pour affiner la compréhension de la structuration du génome d’une bactérie modèle, Escherichia coli.
Ils ont ainsi montré que chaque région activement transcrite du génome adopte une structure tri dimensionnelle spécifique appelée « domaine induit par la transcription » ou TID. Dans une expérience de Hi-C, chaque TID obtient un score qui corrèle directement avec l'activité transcriptionnelle sous-jacente. Ainsi chaque gène ou opéron exprimé va contribuer à la formation d’un mini domaine. Cette observation conduit à réviser les notions de CIDs et de barrières entre CIDs. Les expériences de Hi-C à haute résolution montre que les signaux ayant conduit à leur définition n’étaient que la partie visible d’un signal beaucoup plus général et systématique. Le chromosome consiste donc en une succession de mini domaines transcriptionnels. Leur rôle pour la physiologie de la bactérie est encore inconnu, mais il est envisageable qu’en compartimentalisant ces mini domaines (TIDs), la bactérie maximise l’accès aux ressources nécessaires pour la transcription des gènes
L’activation d’un seul opéron induit la formation d’un TID.
Pour aller plus loin, l'étude utilise un système inductible artificiel pour montrer que l'activation d'une seule unité de transcription (ici un mega opéron de 70 gènes) est suffisante pour générer un TID et cela même si le chromosome est entièrement désorganisé.
Dans cette expérience, les auteurs ont introduit dans le génome de E. coli des promoteurs du bactériophage T7 (un virus bactérien) reconnus par l’ARN polymérase de ce même bactériophage T7. En présence d’un antibiotique, la rifampicine, l’ARN polymérase d’E. coli est inhibée et le génome est entièrement éteint. Or, l’ARN polymérase de T7, elle, est insensible à la rifampicine. Son activation conduit à l’initiation au niveau des promoteurs (segment qui contrôle l’expression d’un gène) T7 de plages de transcriptions en présence de rifampicine, alors que le reste du génome demeure éteint.
Ce système permet ainsi d’induire de manière contrôlée la formation de une, ou deux, plages de transcription sur la totalité du génome, et ainsi d’étudier les propriétés et caractéristiques des TIDs qui en découlent. Cette approche originale a permis de caractériser la nature des TIDs, leurs propriétés d’isolation dans le génome et le surenroulement de l’ADN qui y est associé.
L’étude par imagerie des contraintes mécaniques imposées par les TIDs montrent que ceux-ci influencent la mobilité et la localisation des loci chromosomiques voisins, et que la transcription agit donc comme un acteur fondamental de l'architecture des chromosomes.
Ainsi, cette étude apporte de nouvelles connaissances clés sur la relation dynamique entre transcription et organisation des chromosomes bactériens, en mettant en lumière le rôle des unités transcriptionnelles en tant qu'éléments structurels, ou TIDs, fondamentaux dans ce processus complexe. La transcription étant ubiquitaire chez les êtres vivants, ces résultats ont des implications profondes pour notre compréhension de la dynamique des chromosomes à tous les domaines du vivant.
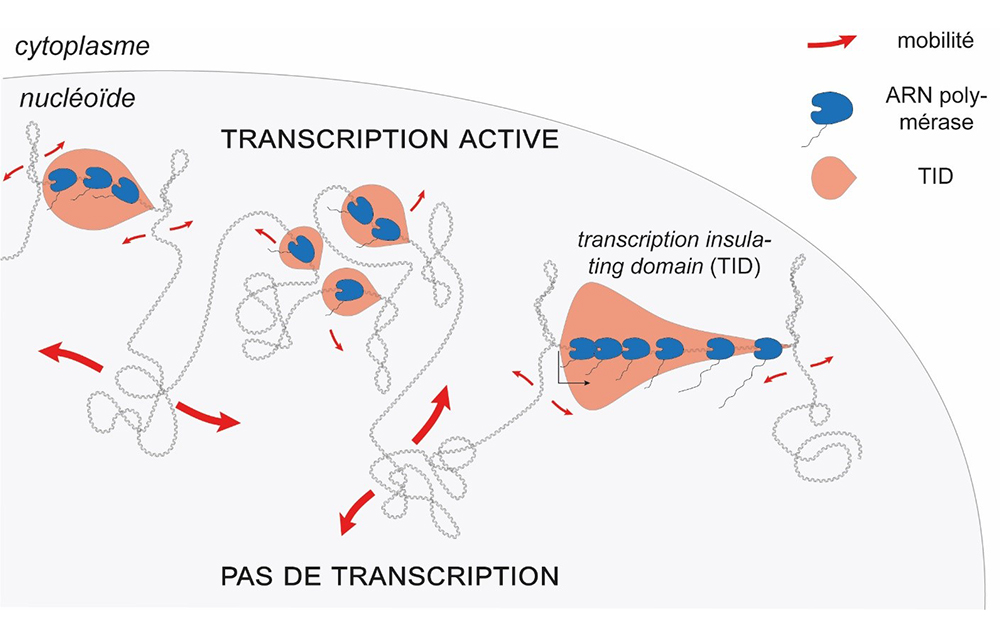
Légende : La transcription dirige le repliement local du chromosome bactérien. Les transcriptions induced domains (TIDs) sont localisés en périphérie du nucléoïde alors que les loci dépourvus de transcription active se situent au centre. La transcription contraint aussi l'ADN en provoquant la formation de surenroulement négatif en aval et positif en amont de l'unité de transcription, ce qui réduit la mobilité de l'ADN.
En savoir plus :
Bignaud, A., Cockram, C., Borde, C. et al. Transcription-induced domains form the elementary constraining building blocks of bacterial chromosomes. Nat Struct Mol Biol (2024). https://doi.org/10.1038/s41594-023-01178-2
Contact
Laboratoires
Génétique des génomes (CNRS/Institut Pasteur)
Institut Pasteur
28 rue du Dr. Roux
75724 Paris Cedex 15
Centre interdisciplinaire de recherche en biologie - CIRB (CNRS/Collège de France/Inserm)
11 place Marcelin Berthelot
75005 Paris
