Cartographier la mécanique d'un embryon d'un seul coup de microscope
Comment mesurer les forces qui sculptent un embryon en développement ? Cette question est restée longtemps un doux rêve pour les scientifiques qui s'intéressent à la morphogenèse, c'est-à-dire à l'émergence des formes biologiques. Dans un article publié dans la revue Nature Methods, les scientifiques proposent une méthode simple et efficace pour cartographier la mécanique embryonnaire.
Le développement des embryons fascine biologistes et physiciens depuis plus d'un siècle. Pourtant les mécanismes génériques qui régissent l'émergence d'une structure complexe et organisée à partir d'une cellule œuf restent encore mal compris. Un embryon se développe généralement selon une séquence plus ou moins invariante d'événements de divisions et réarrangements cellulaires, de signalisation intercellulaire, et de spécification de groupes cellulaires. Pour essayer de déchiffrer cette fascinante chorégraphie, la microscopie en fluorescence est devenue la norme en biologie. Mais peut-elle a priori expliquer, à elle seule, quelles forces sous-jacentes permettent de diriger ce ballet de haute volée ?
La mécanique des cellules joue un rôle important dans la formation des axes de l'organisme
En effet, on sait que les gènes ne font pas tout, et qu'une part importante, voire peut-être essentielle, de l'orchestration spatiotemporelle des formes et brisures de symétrie dans l'embryon sont le fruit d'une mécanique bien huilée. C'est ce que Sir d'Arcy Thompson avait déjà proposé dans son fameux ouvrage "On Growh & Form" au début du 20e siècle, documentant l'analogie frappante entre la forme des cellules dans un embryon de grenouille et l'agencement des bulles de savons confinées entre deux plaques de verre. Depuis, on sait que la mécanique des cellules joue un rôle important dans la formation des futurs axes de l'organisme, et fait partie intégrante des boucles de contrôle dirigeant la formation des tissus primordiaux. Or, les techniques actuelles de mesure de la mécanique à l'échelle cellulaire, telles que l'aspiration par micropipette, l'ablation laser ou l'indentation par microscope à force atomique, sont bien trop lentes ou invasive pour espérer cartographier un embryon entier, et a fortiori en développement.
Analyse d'image, vision par ordinateur et modélisation physique pour obtenir trois dimensions
Il y a quelques années, s'appuyant sur cette analogie formelle entre mousses et tissus, qui partagent une certaine similarité dans leur organisation, l'idée a émergé de pouvoir déduire les tensions relatives aux interfaces cellulaires directement à partir de la géométrie de celles-ci. Ce, via la simple écriture de l'équilibre mécanique, une branche de la physique traitant des forces et des mouvements dans les objets statiques. Cette technique, dite d'inférence de force, a montré son efficacité à deux dimensions, en particulier pour créer des cartes de tensions et pressions cellulaires de tissus épithéliaux, qui forment l’interface entre l'extérieur et l'intérieur de l'organisme. Mais aucune tentative jusque-là de généralisation de la méthode à trois dimensions n'a été très concluante. C'est exactement ce que le travail publié dans la revue Nature Methods, permet désormais de faire, s'appuyant sur une combinaison d'approches d'analyse d'image, de vision par ordinateur et de modélisation physique.
Identifier les interactions qui régissent les tous premiers instants de la vie
Fondamentalement, une difficulté majeure réside dans l'extraction géométrique précise des arrangements cellulaires à partir de la fluorescence des membranes captée par microscopie. Pour ce faire, l'approche s'appuie sur des maillages triangulaires, largement utilisés en ingénierie, et qui sont ici ajustés au mieux à la forme des cellules pour mesurer facilement les angles de contact aux jonctions et les courbures aux interfaces cellulaires. Ce faisant, le problème inverse mathématique consistant à retrouver les tensions cellulaires à partir des angles puis les pressions cellulaires à partir des courbures peut être facilement formulé et résolu.
La méthode a d'abord été évaluée systématiquement grâce à des simulations numériques de mousses cellulaires en trois dimensions, pour être ensuite appliquée à de vrais embryons, en particulier ceux de souris, d'un vers (Caenorhabditis elegans) et d'une ascidie (Phallusia Mammillata, i.e. un animal marin). Les résultats fournissent, pour la première fois, des cartes mécaniques complètes de ces embryons animaux, et ouvrent de nombreuses perspectives, comme celle de généraliser la technique aux tissus végétaux ou de les coupler aux cartes d'expression génétique récemment obtenues grâce aux progrès en séquençage. Cette avancée pourrait, à terme, permettre d'identifier enfin les interactions tenues entre mécanique, signalisation et contrôle génétique qui sous-tendent l'auto-organisation des systèmes multicellulaires et qui régissent les tous premiers instants de la vie.
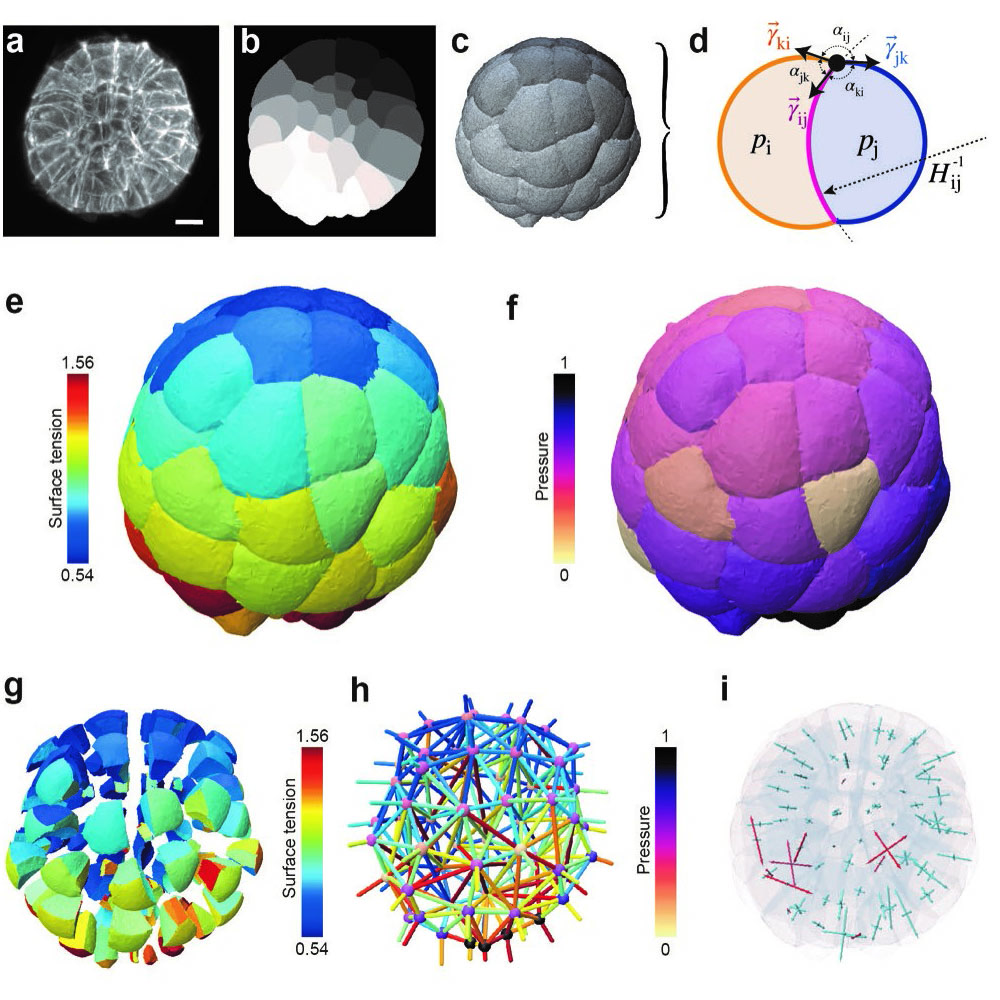
Figure : Inférence de force en 3D. a) Image de microscopie en fluorescence d'un embryon d'ascidie de 64 cellules (source: [4], échelle: 20μm). b) Masque de segmentation dans un plan focal. c) Reconstruction de la surface sous forme d'un maillage. d) Schéma de l'équilibre mécanique sur un doublet cellulaire. e) Carte 3D des tensions relatives dans l'embryon. f) Carte 3D des pressions relatives dans l'embryon. g) Vue explosée de la carte des tensions. h) Graphe de forces (pressions aux nœuds, tensions aux arêtes). i) Carte des contraintes mécaniques diagonalisées par cellule (compressives en bleu, extensibles en rouge).
En savoir plus :
Ichbiah, S., Delbary, F., McDougall, A. et al. Embryo mechanics cartography: inference of 3D force atlases from fluorescence microscopy. Nat Methods 20, 1989–1999 (2023). https://doi.org/10.1038/s41592-023-02084-7
Contact
Laboratoire
Centre interdisciplinaire de recherche en biologie - CIRB (CNRS/Collège de France/Inserm)
11, place Marcelin Berthelot,
75005 Paris