Le bruit des cellules : un mécanisme, deux modèles
Dans les cellules, la lecture de l’information génétique ne se fait pas continûment, mais de manière stochastique et intermittente. Ce phénomène, appelé bruit d’expression, joue un rôle clé dans des nombreuses situations en biologie. Il est responsable du rebond de la charge virale lorsque la trithérapie VIH est arrêtée mais il est aussi incontournable lors du développement embryonnaire. Dans ces deux articles, publiés conjointement dans la revue Nature Communications, les scientifiques ont utilisé le marquage de l’ARN messager et un traitement mathématique novateur pour montrer que la modulation du bruit d’expression dans les cellules infectées avec le VIH et dans les embryons de drosophile utilise un mécanisme commun, la pause proximale au promoteur de l’ARN polymérase Pol II.
Modèle drosophile : En utilisant l’embryon de drosophile comme organisme modèle, il est possible de suivre l’allumage des gènes en temps réel dans des embryons vivants. Ce réveil du génome s’opère de manière discontinue, car la transcription requiert l’assemblage de nombreux acteurs dont l’action est centralisée au niveau d’une séquence d’ADN particulière appelée promoteur. Pour déterminer la contribution des séquences du promoteur à la dynamique de la transcription, les scientifiques ont combiné des manipulations génétiques à de l’imagerie quantitative pour révéler les différents états des promoteurs et leur cinétique in vivo.
Ainsi, ils ont pu mettre en évidence que les promoteurs dits "à boite TATA" sont actifs très précocemment et opéraient via une simple transition entre un état inactif et un état permissif (actif), où une polymérase initie environ toutes les 10 secondes. Pour les promoteurs dits "à motif INR" (Initiateur), actifs plus tardivement, un état inactif supplémentaire existe, celui de la pause de la polymérase. Ces multiples états des promoteurs suggèrent un contrôle de leur activité à différentes échelles temporelles. Dans le contexte d’un embryon en développement rapide, comme lors des étapes des clivages embryonnaires chez les vértébrés et invertébrés, ce contrôle multi-échelle pourrait contribuer à la robustesse de l’expression génique lors du réveil du génome zygotique.
Modèle HIV : Chez les patients infectés par le HIV, il n'est pour l'instant pas possible d'éliminer le virus et les personnes doivent suivre un traitement à vie. L'éradication du virus est impossible à cause de formes latentes du virus, dans lesquelles une cellule infectée n'exprime pas le génome viral et reste indétectable par le système immunitaire. Cependant, ces virus latents peuvent occasionnellement se réveiller et relancer la propagation virale. C'est ce qui produit des rebonds viraux chez les patients qui arrêtent leur thérapie antivirale. Les mécanismes qui permettent le réveil viral dans les cellules latentes font l'objet de nombreuses études. Un des mécanismes principaux qui limite la transcription dans les cellules latentes est l'arrêt des ARN polymérases à proximité du promoteur viral. En effet, cet arrêt, aussi appelé pause, bloque l'entrée de nouvelles polymérases et inhibe ainsi l'expression du virus.
En utilisant une méthode qui permet de visualiser les ARN viraux dans les cellules vivantes avec une sensibilité de l'ordre de la molécule unique, les scientifiques ont analysé la transcription du génome viral dans des conditions qui miment celles des cellules latentes. De manière surprenante, ces cellules montrent de brefs pulses de transcription séparés par de longues périodes d'inactivité de plusieurs heures. Les scientifiques montrent que, contrairement à ce que l'on pensait précédemment, la pause des ARN à proximité du promoteur viral est un évènement stochastique et relativement rare (1/20 à 1/200 des polymérases font cet arrêt). Ainsi, lorsqu'une polymérase arrêtée se débloque, ce qui se produit de temps en temps de manière aléatoire, tout un convoi de nouvelles polymérases peut transcrire le génome viral avant qu'une polymérase ne s'arrête à nouveau. Ceci suffit à produire suffisamment de composants viraux pour infecter de nouvelles cellules et débuter un nouveau cycle infectieux.
Les mathématiques décodent le bruit des cellules
Les scientifiques ont basé le traitement mathématique de ces deux modèles sur une même méthode. Cette méthode génère une carte complète de positions des polymérases (telle une image de toutes les voitures sur une autoroute) pour chaque site de transcription. Ensuite, elle utilise des solutions exactes de ce qu’on appelle en mathématiques un « problème inverse » pour remonter aux taux de transition entre les états moléculaires du promoteur et quantifier ainsi la probabilité et la durée des pauses.
Ainsi, par l'analyse de deux systèmes expérimentaux très différents et l'utilisation d’un traitement mathématique des images "rendant visible l’invisible", les scientifiques démontrent que la pause de la RNA polymérase est facultative et stochastique, contribuant ainsi à l’intermittence de l’expression génétique.
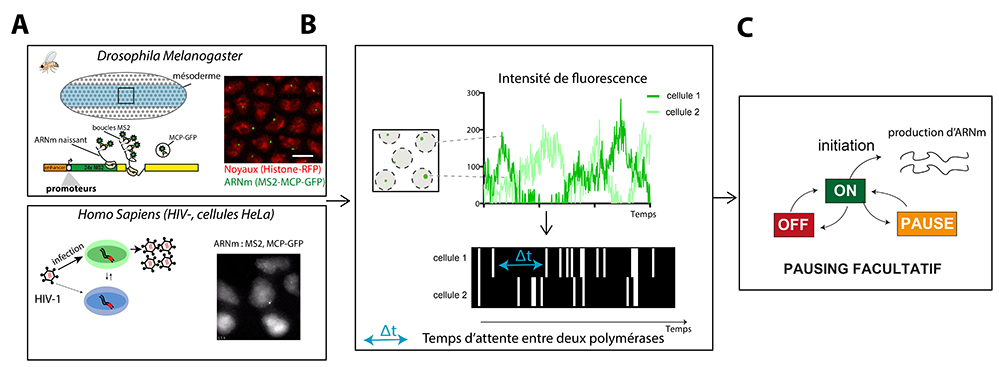
Figure : A-Imagerie de la production d’ARNm dans des cellules vivantes. Les deux études utilisent le système MS2/MCP qui permet de marquer les ARNm (contenant les boucles MS2) par leur liaison avec une RNA binding protéine, la MCP, couplée à de la GFP. Par cette amplification du signal, il est possible de visualiser la synthèse des ARN et de savoir où, quand et avec quelle ‘force’ la transcription s’opère dans des cellules vivantes. L’étude de Pimmett et al., utilise ce système dans des embryons de Drosophile vivants pour analyser l’impact des séquences promotrices sur la dynamique de la transcription. L’étude de Tantale et al., elle, utilise ce même système d’amplification du signal pour visualiser la transcription du virus HIV dans des cellules humaines infectées de manière latente.
B-Déconvolution du signal pour placer les événements d’initiation de la transcription au cours du temps. Le signal fluorescent émanant de chaque noyau est constitué par plusieurs ARN naissants, produits par plusieurs polymérases, initiées chacune à un temps t. Afin de déterminer ces moments précis où chaque polymérase initie la transcription, les chercheurs ont mis en place une méthode innovante de machine learning.
C-Modélisation des étapes cinétiques limitant la transcription. L’analyse statistique des intervalles de temps entre les événements d’initiation de la transcription permet d’estimer le nombre d’étapes limitantes et de déterminer quel modèle de transcription correspond aux données expérimentales. Ainsi, les scientifiques ont démontré que certains promoteurs opèrent par une simple régulation entre un état permissif et un état inactif, tandis que d’autres requièrent une régulation par trois étapes cinétiques clés. C’est le cas des promoteurs régulés par la pause de la polymérase (promoteurs du développement et promoteur HIV). Cependant et contrairement au modèle de pause établit, les données suggèrent un nouveau modèle de pause, un état de pause non obligatoire, que seules une partie des polymérases subissent, de manière peu fréquente.
Pour en savoir plus :
Quantitative imaging of transcription in living Drosophila embryos reveals the impact of core promoter motifs on promoter state dynamics.
Pimmett V, Dejean M, Fernandez C, Trullo A, Bertrand A, Radulescu O and Lagha M
Nature Communications. 23 juillet 2021. https://doi.org/10.1038/s41467-021-24461-6
Stochastic pausing at latent HIV-1 promoters generates transcriptional bursting
Katjana Tantale, Encar Garcia-Oliver, Marie-Cécile Robert, Adèle L'Hostis, Yueyuxiao Yang, Nikolay Tsanov, Rachel Topno, Thierry Gostan, Alja Kozulic-Pirher, Meenakshi Basu, Vera Slaninova, Jean-Christophe Andrau, Florian Mueller, Eugenia Basyuk, Ovidiu Radulescu, Edouard Bertrand
Nature Communications 23 juillet 2021. https://doi.org/10.1038/s41467-021-24462-5
Contact
laboratoire
Institut de Génétique Moléculaire de Montpellier (Université de Montpellier/CNRS)
CNRS-UMR 5535
1919 Route de Mende
34293 Montpellier- Cedex 5 - FRANCE
Laboratory of pathogen host interactions (CNRS, Université de Montpellier)
Place Eugène Bataillon,
34095 Montpellier
Institut de Génétique Humaine (CNRS/Université de Montpellier)
141 rue de la Cardonille
Montpellier - FRANCE