Le complexe protéique PMD/PP7L régule l’expression des gènes et transposons chez Arabidopsis.
De nombreux facteurs épigénétiques régulent l’expression des gènes et répriment les éléments transposables (ETs) afin d’assurer l’intégrité cellulaire. Un crible génétique, suivi d’analyses biochimiques, transcriptomiques et phylogénétiques, a montré que les protéines Plant Mobile Domain (PMD) MAIN, MAIL1 et la phosphatase putative PP7L forment une association protéique conservée chez plusieurs dicotylédones et requise pour la bonne expression des gènes et la répression des ETs chez Arabidopsis. Ces travaux sont publiés dans la revue PLOS Genetics.
Au sein des cellules eucaryotes, l’information génétique est maintenue dans le noyau sous la forme de chromatine, un complexe macromoléculaire composé, entre autres, d’ADN enroulé autour des protéines histones. L’expression des gènes est finement régulée par de nombreux mécanismes moléculaires qui sont interconnectés, ceci afin de permettre la synthèse des protéines nécessaires au bon développement de l’organisme. La première étape de l’expression génique est la transcription de l’ADN en ARN messager. Les mécanismes épigénétiques tels que la méthylation de l’ADN ou les modifications post-traductionnelles des histones jouent un rôle essentiel dans la régulation de la transcription. Celle-ci est aussi régulée par des protéines appelées facteurs de transcription (FTs) qui, selon les cas, vont avoir un rôle d’activateur ou de répresseur transcriptionnel grâce à la reconnaissance de motifs d’ADN spécifiques. En outre, des interactions complexes existent entre mécanismes épigénétiques et FTs ; certaines modifications épigénétiques permettent le recrutement ou l’éviction de FTs particuliers, influant ainsi sur l’expression génique. Finalement, les mécanismes épigénétiques jouent aussi un rôle essentiel dans la répression transcriptionnelle des éléments d’ADN répétés tels que les éléments transposables (ETs) afin de protéger l’intégrité du génome.
Les protéines « Plant Mobile Domain » (PMD) sont des protéines de fonction inconnue qui sont spécifiquement et largement répandues au sein des plantes à fleurs. Bien que la plupart des PMD soient associées à des ETs, les plantes ont domestiqué les PMD pour produire des versions géniques qui jouent des rôles importants au sein de la cellule. Chez la plante modèle Arabidopsis thaliana, « MAINTENANCE OF MERISTEMS » (MAIN) et « MAIN-LIKE 1 » (MAIL1) sont deux protéines PMD impliquées dans les processus développementaux de la plante, la stabilité du génome et la répression transcriptionnelle des ETs. Cependant, les mécanismes impliquant MAIN et MAIL1 dans ces processus cellulaires restent insaisissables.
Les scientifiques ont récemment montré que les mécanismes cellulaires impliquant la protéine MAIN dans la répression des ETs sont indépendant de la méthylation de l’ADN, et que ces deux voies épigénétiques coopèrent pour réprimer les ETs. Par des expériences d’immunoprécipitation couplée à des analyses de spectrométrie de masse, ils ont démontré que les protéines MAIN et MAIL1 interagissent avec la phosphoprotéine phosphatase (PPP) nommée PP7-like (PP7L) pour former le complexe PMD/PP7L qui est requis pour la répression des ETs mais aussi pour la bonne expression de gènes. Ils ont également identifié un motif d’ADN (appelé le motif ‘DOWN’) plus spécifiquement retrouvé au sein de nombreux gènes dont l’expression nécessite le complexe PMD/PP7L fonctionnel.
Des analyses phylogénétiques au sein des dicotylédones ont finalement révélé que les domaines PMD et PP7 sont l'objet d'une co-évolution. Ainsi, plusieurs espèces végétales, dont A. thaliana, expriment des protéines portant un domaine PPP fusionné au domaine PMD. Les chercheurs proposent que l'interaction des domaines PMD et PPP créerait un module protéique fonctionnel impliqué dans des mécanismes de régulation génique et de répression des ETs.
Il reste à déterminer si le complexe PMD/PP7L interagit avec la chromatine et l’ADN, ou bien s’il recrute un FT encore inconnu afin de permettre l’activation transcriptionnelle de gènes via la reconnaissance du motif ‘DOWN’. De même, il faudra élucider les mécanismes moléculaires impliquant le complexe PMD/PP7L dans la répression des ETs et étendre toutes ces analyses aux espèces végétales d'intérêt agronomique.
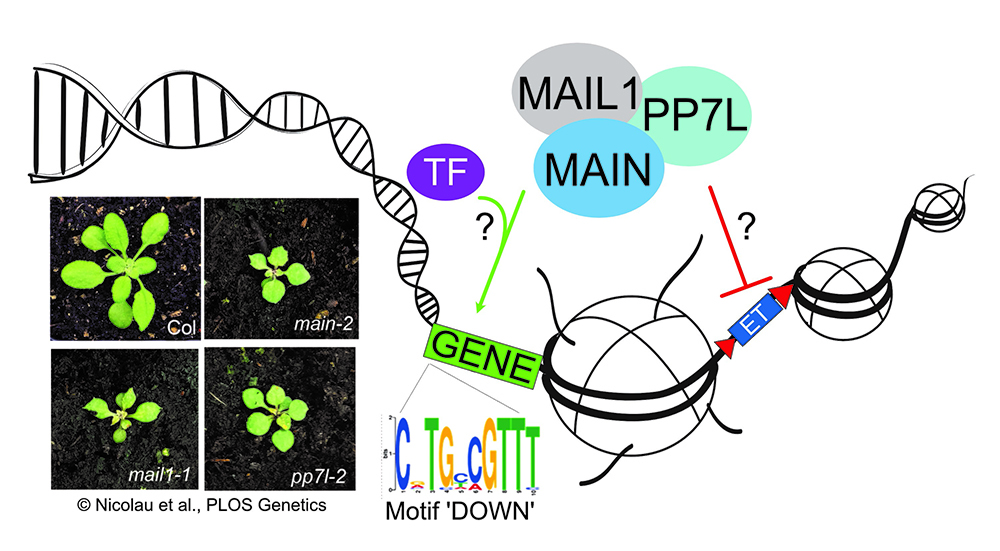
Figure : Les protéines PMD MAIN et MAIL1 s’associent avec la protéine PP7L pour former un complexe qui est requis pour la bonne expression des gènes, la répression des TEs et ainsi le bon développement de la plante. Des plantes mutées pour les gènes MAIN, MAIL1 et PP7L ont des phénotypes développementaux très forts et similaires. De plus, des analyses de comparaison de séquences ont identifié un motif d’ADN (appelé motif ‘DOWN’) qui est enrichi au sein de plusieurs gènes nécessitant la présence du complexe PMD/PP7L pour être exprimés. Dans notre modèle, le complexe PMD/PP7L pourrait directement interagir avec le motif ‘DOWN’ pour permettre l’expression de ces gènes. Il pourrait aussi activer un facteur de transcription (TF) encore non identifié. Finalement, le complexe PMD/PP7L est aussi requis pour la répression des ETs par un mécanisme encore inconnu. Col = écotype Columbia sauvage.
Pour en savoir plus:
The plant mobile domain proteins MAIN and MAIL1 interact with the phosphatase PP7L to regulate gene expression and silence transposable elements in Arabidopsis thaliana.
Nicolau M, Picault N, Descombin J, Jami-Alahmadi Y, Feng S, Bucher E, Jacobsen SE, Deragon JM, Wohlschlegel J, Moissiard G.
PLoS Genet. 2020 Apr 14;16(4):e1008324. doi: 10.1371/journal.pgen.1008324. eCollection 2020 Apr.
Contact
Laboratoire
Laboratoire Génome et Développement des Plantes (LGDP) - (CNRS/UPVD)
Bâtiment T, 58 Avenue Paul Alduy 66860 Perpignan