Un mécanisme inédit d’extension de la recombinaison homologue chez les bactéries
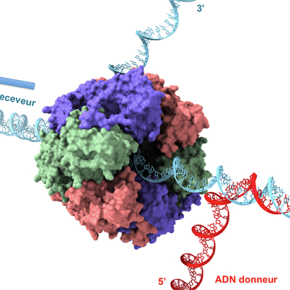
Les étapes précoces de la recombinaison homologue (RH) consistent en l’invasion d’un seul brin d’ADN "donneur" dans un duplex complémentaire "receveur", générant une synapse à trois brins d’ADN communément appelée "D-loop". Les équipes de Patrice Polard au Laboratoire de microbiologie et de génétique microbiennes, et Rémi Fronzes à l’Institut européen de chimie et de biologie, dévoilent un mécanisme inédit d’extension de l’incorporation d’ADN au niveau de la D-loop de RH. Cette étude a été publiée le 31 mai 2017 dans la revue Nature Communications.
Les recombinases RecA/Rad51 sont des effecteurs centraux des étapes d’échanges de brins d’ADN de multiples voies de recombinaison homologue (RH) essentielles pour la la stabilité et l’évolution des génomes chez tous les organismes. Elles catalysent la RH sous la forme de polymères assemblés et désassemblés de manière ordonnée sur les brins d’ADN échangés, un processus régi par la fixation et l’hydrolyse d’ATP à l'interface de chaque monomère du filament. La RH débute par la polymérisation de la recombinase sur un brin d’ADN, générant un nucléofilament actif pour son appariement à une séquence d’ADN double-brin (ADNdb) homologue. Il en résulte un intermédiaire à 3 brins d’ADN, communément appelé "D-loop".
Les voies de RH se distinguent entre elles par des effecteurs secondaires, qui contrôlent ou assistent l’action de la recombinase. Singulièrement, plusieurs de ces effecteurs présentent une homologie significative de séquence avec les protéines RecA/Rad51 au niveau du site de liaison à l’ATP. Leur rôle, peu compris jusqu’à présent sur le plan du mécanisme, est déterminant pour la physiologie cellulaire. En effet, des mutations dans les 5 protéines humaines de ce type entrainent une forte susceptibilité à développer des cancers.
L’appareil de RH bactérien possède une seule protéine de ce type. Il s’agit de la protéine fortement conservée au plan évolutif RadA (aussi appelée Sms dans certaines espèces), identifiée et caractérisée génétiquement il y a une trentaine d’années chez Escherichia coli pour son rôle d’assistance à RecA dans la réparation de dommages à l’ADN du génome. Son étude biochimique a récemment montré qu’elle agit en soutien à RecA pour promouvoir l’incorporation d’ADN simple-brin (ADNsb) au niveau de la D-loop. L’homologie entre RadA et RecA a suggéré que RadA mimerait et/ou assisterait le mécanisme de migration de branche d’ADN de RecA, qui catalyse au niveau de la D-loop l’incorporation de l’ADNsb envahissant, de manière biaisée vers son extrémité 3’.
Les chercheurs ont conduit une étude de la structure et de la fonction de la protéine RadA de la bactérie pathogène de l’homme Streptococcus pneumoniae. Cette étude intégrée a révélé sa structure atomique. De manière inattendue, son domaine central apparenté à RecA a montré sa très forte paralogie avec le domaine de liaison et d’hydrolyse de l’ATP des hélicases réplicatives bactériennes de la famille DnaB. A cette découverte s’ajoute l’organisation de RadA en anneau hexamérique, un trait commun aux protéines DnaB. Ceci a guidé l’étude biochimique de RadA, qui a révélé qu’elle était une hélicase active se déplaçant comme DnaB le long de l’ADNsb de 5’ vers 3’. Une autre avancée de cette caractérisation fonctionnelle de RadA du pneumocoque a résulté de son étude in vivo visant à comprendre son rôle dans la transformation génétique, un processus de transfert latéral d’ADN conduisant à son intégration au génome par RH. L’inactivation de RadA conduit à une réduction de l’efficacité de transformation de 100 fois. A l’aide de tests de transformation particuliers, les chercheurs ont montré que le rôle de RadA est de promouvoir l’intégration d’ADNsb dans le génome sur de longues distances, dans la direction 3’ de cet ADNsb recombiné, mais aussi dans la direction 5’, à l’opposé de l’action de migration de branche catalysée par RecA. L’interaction de RadA avec RecA, également révélée dans cette étude, est nécessaire à cette action de RadA.
Cette étude aboutit à un modèle de mécanisme totalement inédit d’assistance de la RH médiée par une hélicase de type DnaB. Ce modèle, réconciliant les études biochimiques et génétiques, propose qu’un hexamère de RadA serait chargé par RecA sur chaque brin de l’ADNdb receveur et prolongerait symétriquement l’incorporation d’ADNsb donneur aux bornes de la D-loop construite par RecA. RadA émerge comme un effecteur d’extension de l’appareil de recombinaison homologue bactérien, une activité optimisant la plasticité du génome lors de la transformation génétique.
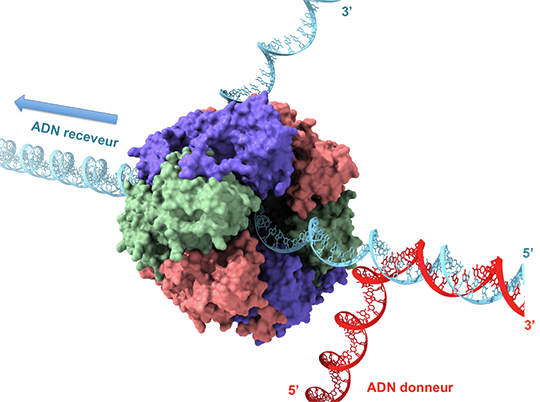
© Rémi Fronzes
En savoir plus
-
Bacterial RadA is a DnaB-type helicase interacting with RecA to promote bidirectional D-loop extension.
Marie L, Rapisarda C, Morales V, Bergé M, Perry T, Soulet AL, Gruget C, Remaut H, Fronzes R, Polard P.
Nat Commun. 2017 May 31;8:15638. doi: 10.1038/ncomms15638