Echange d'armes dans la bataille entre les éléments transposables et leur hôte
De multiples mécanismes épigénétiques assurent l'inactivation des éléments transposables au sein des génomes. L'équipe d'Olivier Mathieu, au laboratoire Génétique, reproduction et développement, identifie deux gènes nécessaires à la répression de nombreux éléments transposables, via une nouvelle voie moléculaire indépendante des principales modifications épigénétiques. Ces gènes pourraient avoir eux-mêmes dérivé d'éléments transposables. Ceci révèle que la domestication d’éléments transposables par les génomes des plantes à fleurs aurait permis l'émergence d'un nouveau mécanisme de contrôle de l'activité de ces séquences invasives. Cette étude a été publiée le 3 mai 2017 dans la revue Nature Communications.
Les éléments transposables sont des séquences d'ADN mobiles, capables de se dupliquer au sein des génomes en s'intégrant à de nouvelles positions chromosomiques. Ces éléments représentent une fraction importante du génome de la majorité des êtres vivants et contribuent de manière significative aux variations de taille observées entre les génomes; ainsi, les séquences dérivées d’éléments transposables constituent environ 50% du génome humain, une fraction qui peut atteindre plus de 80% pour certains génomes de plantes comme celui du maïs. Si les éléments transposables contribuent largement à l'évolution des génomes, leur caractère mutagène potentiellement délétère a conduit à la mise en place de multiples mécanismes épigénétiques qui permettent au génome hôte de contrôler strictement l'activité transcriptionnelle de ces éléments.
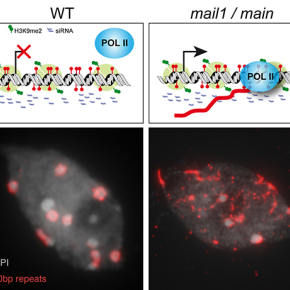
Les marques épigénétiques englobent les modifications chimiques, réversibles, qui sont apposées sur la chromatine (bases de l'ADN et histones) et peuvent être transmises de manière stable lors des divisions cellulaires. Ainsi, de nombreuses voies de régulation épigénétique contrôlent l'expression des éléments transposables via l'imposition et le maintien sur ces éléments d'un fort niveau de méthylation de l'ADN. Dans la plante modèle Arabidopsis thaliana, ce ciblage dépend en partie de petits ARN interférents et une forte corrélation existe entre cette hyperméthylation de l’ADN et d'autres modifications épigénétiques touchant les histones comme la méthylation de la lysine 9 (H3K9me2) ou de la lysine 27 (H3K27me1) de l'histone H3. La juxtaposition de ces marques est associée à la mise en place de structures hétérochromatiques "fermées" dans lesquelles la chromatine fortement condensée est peu propice à la transcription. L'archétype de cette structure compacte est visible dans les régions péricentromériques des chromosomes où est localisée une large fraction des éléments transposables du génome.
Par une approche génétique, l'équipe d'Olivier Mathieu met en évidence une nouvelle voie de régulation, contribuant notamment à l'inactivation des éléments transposables. La dérégulation transcriptionnelle observée dans le mutant kumonosu (kun) n'est pas tributaire des petits ARN interférents et elle est largement indépendante de la méthylation ADN et des autres marques épigénétiques communément associées à l'hétérochromatine. Dans cette étude, les chercheurs montrent que le phénotype kun est lié à une mutation unique dans le gène MAIL1 et qu'un gène fortement homologue, MAIN, est impliqué dans ce mécanisme d’inactivation transcriptionnelle ou « silencing », probablement via la formation d'hétérodimères MAIL1-MAIN. Bien que le mécanisme moléculaire de la voie MAIL1/MAIN reste à définir, ce complexe apparaît nécessaire à la condensation de l'hétérochromatine. En l'absence d'un des 2 facteurs identifiés par cette étude, on observe une désorganisation structurale et fonctionnelle des régions péricentromériques, associée à une réactivation transcriptionnelle de nombreux éléments transposables.
Les protéines MAIL1/MAIN contiennent un seul domaine identifiable, appelé "Plant Mobile Domain". L'étude évolutive de ce domaine, menée en collaboration avec Jean-Marc Deragon (Laboratoire génome et développement des plantes, Perpignan), suggère qu'il était à l'origine présent au sein d'éléments transposables et qu'il a ensuite été "capturé" par le génome hôte pour son propre profit. Cet événement de domestication a eu lieu chez l'ancêtre commun des plantes à fleurs et a conduit paradoxalement à l'émergence d'une nouvelle classe de gènes dont la fonction vise à restreindre l'autonomie des éléments transposables. Ces données soulignent l'extraordinaire capacité du génome à s'adapter et à développer de nouvelles stratégies pour enrayer l'invasion et contrôler la prolifération d'éléments potentiellement délétères.
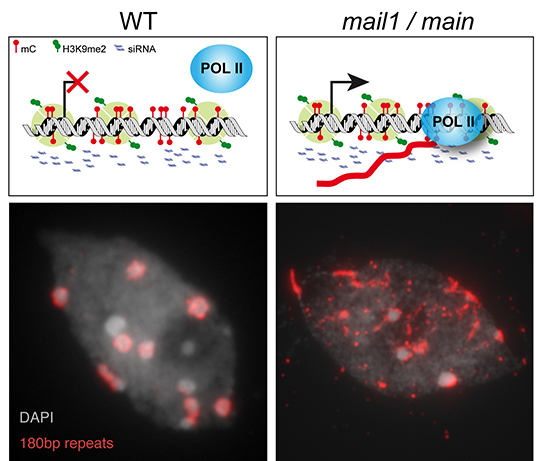
© Olivier Mathieu
En savoir plus
-
Arabidopsis proteins with a transposon-related domain act in gene silencing.
Ikeda Y, Pélissier T, Bourguet P, Becker C, Pouch-Pélissier MN, Pogorelcnik R, Weingartner M, Weigel D, Deragon JM, Mathieu O.
Nat Commun. 2017 May 3;8:15122. doi: 10.1038/ncomms15122.