Comment ajuster la biogénèse d’un complexe multiprotéique
Si de nombreuses activités cellulaires mettent en jeu des édifices protéiques formés de plusieurs sous-unités, les régulations contrôlant leur homéostasie restent mal connues. Des chercheurs de l’Institut Jacques Monod ont utilisé le complexe du pore nucléaire (NPC) comme paradigme pour déchiffrer les mécanismes qui assurent la biogénèse de tels assemblages, et maintiennent leur stœchiométrie. Leurs travaux, publiés le 25 avril 2018 dans la revue Nature communications, révèlent une boucle de régulation par laquelle la biogénèse des sous-unités du NPC est ajustée en fonction de l’intégrité du complexe.
L’identité et le fonctionnement des cellules reposent de manière déterminante sur la formation de polymères de biomolécules. Le maintien de l’architecture cellulaire et la réalisation de processus enzymatiques mettent notamment en jeu des assemblages supramoléculaires formés par de multiples polypeptides. Pour assurer la biogénèse de tels complexes, les différentes étapes de l’expression des gènes synchronisent la synthèse des différentes sous-unités protéiques, et évitent ainsi l’accumulation de polypeptides « orphelins », susceptibles d’interférer avec l’assemblage ou l’activité du complexe entier. Cependant, dans la plupart des cas, on ignore encore les mécanismes qui permettent de contrôler l’expression de ces sous-unités selon leur stœchiométrie, et de l’adapter aux besoins de la cellule en fonction de son état physiologique.
Ce sont ces régulations géniques qu’a cherché à comprendre une équipe de l’Institut Jacques Monod, en collaboration avec le centre d’Epigénétique et Destin Cellulaire (UMR 7216 CNRS, Université Paris Diderot) et le laboratoire de Biologie Computationnelle et Quantitative (UMR 7238 CNRS, Sorbonne Université). L’étude a été plus particulièrement axéesur le pore nucléaire (NPC), un complexe multiprotéique très conservé, formé d’une trentaine de sous-unités distinctes, les nucléoporines, elles-mêmes présentes en plusieurs copies par pore.
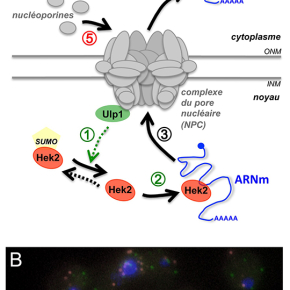
Les chercheurs ont exploité des analyses génomiques décrivant l’association de différentes protéines régulatrices à leurs ARNm cibles chez la levure Saccharomyces cerevisiae et montré qu’une fraction des transcrits codant pour ces nucléoporines est spécifiquement associée avec une protéine nommée Hek2, apparentée aux hnRNP E/K, des protéines de liaison aux ARN impliquées dans des régulations post-trancriptionnelles. Ils ont également pu établir que la répression traductionnelle exercée par Hek2 sur ces ARNm, ainsi que l’activité du protéasome (complexe en charge de la dégradation des protéines), concourent à limiter l’accumulation et l’agrégation de nucléoporines non-assemblées. Ce mécanisme est en outre réversible et contrôlé en réponse à une demande accrue de nucléoporines. Pour cela, la protéine Hek2 est couplée de manière covalente au polypeptide SUMO (Small Ubiquitin-like Modifier, responsable de régulations post-traductionnelles) et cette modification inhibe son activité de liaison aux ARNm, autorisant donc leur traduction en nucléoporines. De plus, des défauts d’intégrité des NPCs, observés dans différentes situations mutantes ou physiologiques, affectent l’activité d’Ulp1, une SUMO-protéase associée aux pores, ce qui entraine l’accumulation de molécules Hek2 SUMOylées, donc incompétentes pour lier leurs ARNm cibles.
Le modèle, illustré dans la figure, propose que la SUMO-protéase Ulp1 et le répresseur traductionnel Hek2 soient respectivement senseur et effecteur d’un mécanisme maintenant l’intégrité de ces complexes. On peut noter que chez les mammifères, de manière comparable, certains ARNm codant pour des nucléoporines sont liés par la protéine hnRNP K, apparentée à Hek2, même si les conséquences de cette association restent à déterminer. La protéine hnRNP K est également couplée à SUMO, cette modification étant contrôlée par une SUMO-protéase associée aux NPCs, suggérant une conservation évolutive de ces mécanismes de régulation.
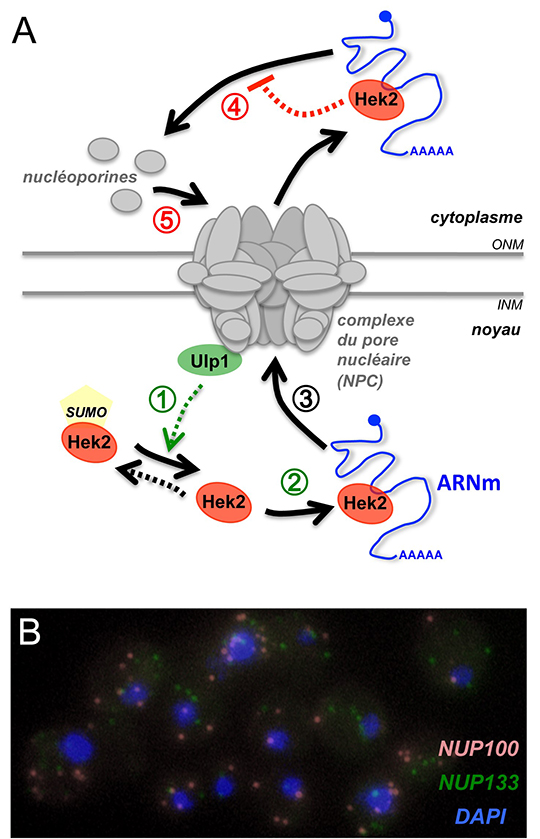
© Benoit Palancade
En savoir plus
-
A SUMO-dependent feedback loop senses and controls the biogenesis of nuclear pore subunits.
Rouvière JO, Bulfoni M, Tuck A, Cosson B, Devaux F, Palancade B.
Nat Commun. 2018 Apr 25;9(1):1665. doi: 10.1038/s41467-018-03673-3.